شروع کار با PyGamLab V2
این ابزار برای موارد زیر فراهم میکند:
ساخت و نمایش ساختارهای اتمی و نانوساختارها
اتصال به پایگاههای داده مواد
بارگذاری و تنظیم دقیق مدلهای هوش مصنوعی از پیش آموزش دیده
تحلیل دادههای تجربی
استفاده از ثوابت بنیادی، ابزارهای تبدیل واحد و فرمولهای مهندسی
این راهنما به شما کمک میکند تا PyGamLab را نصب کنید و ابزارها، توابع و کلاسهای آن را گام به گام بررسی کنید. میتوانید آموزش را از ابتدا تا انتها دنبال کنید یا با کلیک روی بخشهای فهرست مطالب، مستقیماً به موضوع مورد نظر بروید.
فهرست مطالب¶
۰. نمای کلی PyGamLab
۱. نحوه نصب
۲. ثوابت و تبدیلکنندهها
۳. توابع
۴. نانوساختارها
۵. پایگاههای داده
۶. تحلیل دادههای نانو
۷. هوش مصنوعی نانو – مدلهای از پیش آموزش دیده
۸. ضمیمه الف: کار با NumPy
۹. ضمیمه ب: کار با Pandas
۱۰. ضمیمه ج: کار با scikit-learn
۰. نمای کلی PyGamLab¶
PyGamLab یک کتابخانه جامع پایتون است که برای توانمندسازی محققان، مهندسان و دانشجویان فعال در علوم مواد و فناوری نانو طراحی شده است. این کتابخانه محاسبات علمی، هوش مصنوعی و ابزارهای تخصصی را ترکیب میکند تا فرآیندهای کاری از تحلیل داده تا شبیهسازی و مدلسازی را سادهسازی کند.
در این بخش، نمای کلی دقیقی از قابلیتهای اصلی PyGamLab و نحوه پشتیبانی از تحقیقات و پروژههای شما ارائه میدهیم.
ویژگیهای کلیدی¶
این ویژگی به ویژه برای آمادهسازی شبیهسازی، طراحی نانوساختار و اهداف آموزشی مفید است.
این قابلیت جمعآوری دستی داده را کاهش میدهد و فرآیند کاری شما را سریعتر و قابل تکرارتر میکند.
ماژول هوش مصنوعی به شما امکان میدهد از یادگیری ماشین پیشرفته بدون نیاز به پیادهسازی مدلها از ابتدا استفاده کنید.
این ابزارها برای علوم نانو و دادههای مواد طراحی شدهاند و بینشهای سطح بالا را با حداقل کد ارائه میدهند.
این ابزارها کدهای تکراری را کاهش میدهند و اطمینان میدهند که محاسبات دقیق و سازگار هستند.
این آموزش چگونه به شما کمک میکند¶
در این آموزش، تمام ماژولهای اصلی PyGamLab را با مثالهای عملی مرور خواهیم کرد:
شروع با نصب و تنظیم.
بررسی ثوابت، تبدیلکنندهها و توابع کاربردی.
یادگیری کار با مجموعه دادهها با استفاده از توابع و ابزارهای تحلیل.
ساخت و نمایش نانوساختارها برای شبیهسازی.
استفاده از مدلهای هوش مصنوعی برای پیشبینی و تحلیل پیشرفته داده.
در پایان این آموزش، درک روشنی از قابلیتهای PyGamLab و نحوه اعمال آنها در پروژههای تحقیقاتی خود یا دورههای آموزشی در علوم مواد و فناوری نانو خواهید داشت.
۱. نحوه نصب¶
قبل از استفاده از ابزارها، کلاسها و توابع قدرتمند PyGamLab، باید کتابخانه را نصب کنید. مانند اکثر کتابخانههای پایتون، PyGamLab را میتوان با استفاده از pip، مدیر بسته پایتون نصب کرد.
گام ۱: باز کردن رابط خط فرمان¶
ویندوز: کلیدهای
Win + Rرا فشار دهید،cmdرا تایپ کنید و Enter را فشار دهید تا خط فرمان باز شود.MacOS:
Terminalرا از Applications → Utilities باز کنید.لینوکس: برنامه ترمینال مورد نظر خود را باز کنید.
پس از باز کردن ترمینال، میتوانید با نصب ادامه دهید.
گام ۲: نصب PyGamLab¶
میتوانید آخرین نسخه پایدار PyGamLab را با استفاده از دستور زیر نصب کنید:
pip install pygamlab
یا اگر میخواهید آخرین نسخه توسعه را مستقیماً از GitHub دریافت کنید:
pip install git+https://github.com/APMaii/pygamlab.git
نکته: توصیه میشود برای جلوگیری از تداخل با سایر بستههای پایتون از محیط مجازی استفاده کنید. میتوانید یکی با دستور زیر ایجاد کنید:
python -m venv myenv
source myenv/bin/activate # در MacOS/Linux
myenv\Scripts\activate # در ویندوز
گام ۳: تأیید نصب¶
پس از نصب، بررسی کنید که PyGamLab به درستی نصب شده است با اجرای:
import pygamlab
print(pygamlab.__version__)
اگر این دستور شماره نسخه را بدون خطا چاپ کرد، همه چیز آماده است و میتوانید استفاده از PyGamLab را شروع کنید.
نصب pip (در صورت نیاز)¶
اگر هنوز pip را نصب نکردهاید، میتوانید آن را با استفاده از دستور زیر نصب کنید (نیاز به Python 3.4+ دارد):
python -m ensurepip --upgrade
همچنین میتوانید راهنمای رسمی نصب pip را اینجا دنبال کنید: https://pip.pypa.io/en/stable/installation/
پس از نصب pip، میتوانید گام ۲ را دنبال کنید تا PyGamLab را نصب کنید.
۲. ثوابت و تبدیلکنندهها¶
یکی از ایدههای اصلی PyGamLab V1 ارائه مکانی متمرکز برای ثوابت و تبدیلکنندههای واحد بود. این به محققان، مهندسان و دانشجویان کمک میکند تا از جستجوی مکرر مقادیر یا کدگذاری سخت "اعداد جادویی" در کد خود اجتناب کنند.
چرا ثوابت و تبدیلکنندهها مهم هستند¶
ثوابت: اینها متغیرهای از پیش تعریف شدهای هستند که مقادیر مهم فیزیکی، شیمیایی و مهندسی را نشان میدهند. در PyGamLab، ثوابت شامل موارد زیر هستند:
ثوابت فرمول
خواص مواد
خواص نیمههادی
خواص حرارتی، الکتریکی و مکانیکی
با استفاده از ثوابت نامگذاری شده به جای اعداد خام، کد شما خوانایی، نگهداری و اشکالزدایی بهتری پیدا میکند.
تبدیلکنندهها: PyGamLab مجموعهای از توابع برای تبدیل واحدها بین سیستمهای مختلف ارائه میدهد. این به شما امکان میدهد بدون نیاز به محاسبه دستی تبدیلها در هر بار، با واحدهای اندازهگیری مختلف کار کنید.
نحوه پیادهسازی PyGamLab¶
همه ثوابت در ماژول Constants ذخیره شدهاند، در حالی که توابع تبدیل
در توابع کاربردی گروهبندی شدهاند. این طراحی اطمینان میدهد که:
شما هرگز نیاز به حفظ کردن مقادیر یا جستجوی مکرر آنها ندارید.
کد شما خودتوضیحدهنده است، زیرا هر ثابت دارای یک نام معنیدار است.
میتوانید به سرعت واحدها را تغییر دهید در محاسبات، و این باعث میشود آزمایشات یا شبیهسازیهای شما سازگار و دقیق باشند.
مثال استفاده¶
from pygamlab.Constants import PhysicalConstants
from pygamlab.converters import unit_convert
# دسترسی به یک ثابت
speed_of_light = PhysicalConstants.c
print(speed_of_light) # 299792458 m/s
# تبدیل واحدها
length_in_cm = unit_convert(1, from_unit="m", to_unit="cm")
print(length_in_cm) # 100
با این رویکرد، PyGamLab اطمینان میدهد که کد شما تمیز، قابل فهم و از نظر علمی دقیق است.
۳. توابع¶
ماژول Functions در PyGamLab V1 یکی از مهمترین ماژولها برای محققان، مهندسان و دانشجویان در علوم مواد، فناوری نانو، ترمودینامیک و فیزیک است.
ماژول Functions چیست؟¶
ماژول Functions اساساً یک مجموعه از فرمولهای علمی آماده استفاده است. به جای کدگذاری دستی معادلات در هر بار، میتوانید از این توابع برای انجام محاسبات مربوط به موارد زیر استفاده کنید:
خواص مواد
فرآیندهای ترمودینامیکی
بلورشناسی
الکترواستاتیک
پدیدههای مقیاس نانو
این ماژول مجموعهای از فرمولهای قابل اعتماد و تست شده را ارائه میدهد که به طور مکرر در محاسبات علمی استفاده میشوند.
هدف ماژول Functions¶
- صرفهجویی در زمان و کاهش خطاهابه جای پیادهسازی فرمولها خودتان، میتوانید به توابع از پیش ساخته شده و تست شده تکیه کنید. این اشتباهات کدنویسی را کاهش میدهد و محاسبات دقیق را تضمین میکند.
- بهبود خوانایی و قابلیت نگهداریاستفاده از نامهای توصیفی توابع مانند
Coulombs_LawیاActivation_Energyاز "اعداد جادویی" در کد شما جلوگیری میکند و آن را آسانتر برای درک و اشکالزدایی میسازد. - فعالسازی نمونهسازی سریعمیتوانید به سرعت این توابع را در شبیهسازیها یا آزمایشات خود ادغام کنید بدون نگرانی در مورد ریاضیات زیربنایی.
- سازگاری در پروژههابا استفاده از مجموعه استاندارد توابع، محاسبات شما سازگار و قابل تکرار در پروژهها یا همکاران مختلف باقی میماند.
مزایای استفاده از ماژول Functions¶
دقیق از نظر علمی: همه فرمولها بر اساس اصول علمی استاندارد هستند.
خوب مستندسازی شده: هر تابع با توضیحات پارامترها و مقادیر بازگشتی همراه است.
قابل تعامل: به طور یکپارچه با ماژولهای Constants، Converters و AI PyGamLab کار میکند.
چندکاره: توابع طیف وسیعی از حوزهها را پوشش میدهند، از فیزیک کلاسیک تا خواص نانومواد.
مثالهای توابع و استفاده¶
تابع Activation Energy انرژی مورد نیاز برای رخ دادن یک واکنش را با استفاده از معادله آرنیوس محاسبه میکند:
import math
from pygamlab.functions import Activation_Energy
k = 0.01 # rate constant at T
k0 = 1.0 # pre-exponential factor
T = 300 # temperature in Kelvin
Ea = Activation_Energy(k, k0, T)
print(f"Activation Energy: {Ea:.2f} J/mol")
The Bragg Law function calculates the diffraction angle of X-rays through a crystal lattice:
from pygamlab.functions import Bragg_Law
theta = Bragg_Law(h=1, k=1, l=1, a=0.5, y=0.154)
print(f"Diffraction angle: {theta:.2f}°")
This is useful for analyzing crystal structures or interpreting X-ray diffraction data.
The Debye Temperature function estimates the characteristic temperature of a solid material:
from pygamlab.functions import Calculate_Debye_Temperature
theta_D = Calculate_Debye_Temperature(velocity=5000, atomic_mass=63.55, density=8960, n_atoms=1)
print(f"Debye Temperature: {theta_D} K")
The Coulombs_Law function calculates the electrostatic force between two charges:
from pygamlab.functions import Coulombs_Law
F = Coulombs_Law(charge1=1e-6, charge2=2e-6, distance=0.01)
print(f"Electrostatic Force: {F:.4f} N")
Useful for nanostructure simulations, charge interaction analysis, and electrostatic modeling.
The Functions module is a core part of PyGamLab designed to make scientific computations faster, safer, and more readable. By providing a collection of well-documented formulas, this module allows users to focus on research and experimentation rather than coding routine equations. It works best when combined with Constants and Converters, forming a powerful toolkit for materials science and nanotechnology research.
۴. نانوساختارها¶
در قلب PyGamLab V2 ماژول Structures قرار دارد که به طور خاص برای فناوری نانو و علوم مواد محاسباتی طراحی شده است. این ماژول ابزارهایی برای ایجاد، دستکاری، نمایش و صادرات ساختارهای اتمی و در مقیاس نانو ارائه میدهد.
با این ماژول میتوانید تولید کنید:
مواد ۰D (مثلاً خوشهها یا نانوذرات)
مواد ۱D (مثلاً نانوسیمها)
مواد ۲D (مثلاً ورقههای گرافن، تکلایهها)
ساختارهای توده (مواد بلوری، شبکهها)
هر ساختار به عنوان یک کلاس شیگرا نمایش داده میشود که در آن هر اتم، پیوند یا واحد دارای ویژگیها (مثلاً موقعیت، نوع، بار) و متدها (مثلاً انتقال، چرخش، پیوند، محاسبه فاصلهها) است. این امکان کنترل کامل بر ساخت، تغییر و تحلیل ساختارها را فراهم میکند.
پس از ایجاد، ساختارها میتوانند:
تنظیم و دستکاری شوند
با استفاده از چندین موتور نمایش داده شوند
به فرمتهای رایج برای سایر بستهها مانند ASE یا Pymatgen تبدیل شوند
برای ورود به نرمافزارهای شبیهسازی صادر شوند
مزایای ماژول Structures¶
طراحی شیگرا: هر اتم یا پیوند یک نمونه کلاس با ویژگیها و متدها است.
پشتیبانی از تمام ابعاد: ساخت مواد توده 0D، 1D، 2D یا 3D.
ادغام با سایر ابزارها: تبدیل ساختارها به اشیاء ASE یا Pymatgen.
ژنراتورهای قدرتمند: ایجاد ساختارهای پیچیده با خطوط کد کمتر نسبت به نوشتن همه چیز به صورت دستی.
پشتیبانی از نمایش: چندین موتور مانند Matplotlib، PyVista، یا نرمافزار داخلی برای نمایش در زمان واقعی.
مدیریت قوی ساختار: شامل خوانندهها، صادرکنندهها، تبدیلکنندهها و بررسیکنندهها برای مدیریت فایلها و فرمتهای شی.
نمای کلی زیرماژولها¶
زیرماژول PrimAtom واحدهای بنیادی ساختارها را تعریف میکند:
GAM_Atom: نمایش یک اتم.GAM_Bond: نمایش یک پیوند بین اتمها.
اینها بلوکهای سازنده برای همه ساختارها هستند. با چندین
شیء GAM_Atom و GAM_Bond میتوانید نانوساختارهای پیچیده بسازید.
زیرماژول Generators کلاسهای سطح بالا ساخته شده روی اشیاء ASE را ارائه میدهد که به شما امکان میدهد:
ساختارهای پیچیدهای ایجاد کنید که در غیر این صورت بیش از 40 خط کد ASE نیاز دارد.
چیدمانهای اتمی و الگوهای شبکه را به طور خودکار تولید کنید.
عملیات روی ساختار پس از ایجاد انجام دهید.
این به ویژه برای طراحی پیشرفته مواد و تنظیم شبیهسازی مفید است.
زیرماژول GAM_Architecture بر ساختارهای ریاضی و ساخته شده از ابتدا تمرکز دارد. این به شما امکان میدهد:
ساختارها را از ابتدا بسازید (مثلاً گرافن، نانولولهها).
از الگوهای هندسی از پیش تعریف شده برای مواد رایج استفاده کنید.
این پایه ماژول structures است.
زیرماژول I/O موارد زیر را مدیریت میکند:
خواندن: بارگذاری ساختارها در فرمت
GAM_Atom.صادرات: ذخیره ساختارها برای نرمافزارهای خارجی یا شبیهسازیها.
تبدیل: تبدیل بین اشیاء ASE، Pymatgen یا GAM_Atom.
بررسیکننده: شناسایی نوع و اعتبار اشیاء ساختار شما.
زیرماژول Molecular_Visualized (از GAMVIS) امکان نمایش ساختارها را با استفاده از چندین موتور فراهم میکند:
Matplotlib – رسم پایه
PyVista – نمایش سهبعدی تعاملی
موتور داخلی – باز کردن نرمافزار اختصاصی نمایش برای کاوش در زمان واقعی
مثال استفاده¶
#Creation of Au nano cluster
#first from Generators import Nano_OneD_Builder claas
from PyGamLab.structures.Generators import Nano_ZeroD_Builder
#create one object from this object with specific parameters
builder=Nano_ZeroD_Builder(material="Au",
structure_type="nanocluster",
size=3, # or use noshells=3
noshells=3,
crystal_structure="fcc",
lattice_constant=4.08)
#now you have your object and you can utilize its methods
#you can add defects with add_defects method
#you can create alloys and .....
#but for now , juts you can get the atoms (which are from Primatom class)
my_atoms=builder.get_atoms()
#for visulization you can use Molecular_Visulizer class
from PyGamLab.structures.gamvis import Molecular_Visualizer
Molecular_Visualizer(my_atoms,format='efficient_plotly')
#also you can use other formats like 'ase', 'gamvis' and 'pyvista'
Visualizing 55 atoms using efficient_plotly format...
Molecular_Visualizer(my_atoms,format='pyvista')
Visualizing 55 atoms using pyvista format...
/Users/apm/anaconda3/envs/DL/lib/python3.10/site-packages/pyvista/jupyter/notebook.py:56: UserWarning:
Failed to use notebook backend:
No module named 'trame'
Falling back to a static output.

Molecular_Visualizer(my_atoms,format='gamvis')
Visualizing 55 atoms using gamvis format...
=== PyGAMLab Visualizator Debug Info ===
Python version: 3.10.13 | packaged by conda-forge | (main, Dec 23 2023, 15:35:25) [Clang 16.0.6 ]
PyQt5 version: 5.15.2
Platform: Darwin 24.3.0
Creating main window...
Loading 55 atoms...
Showing window...
Initializing VTK interactor...
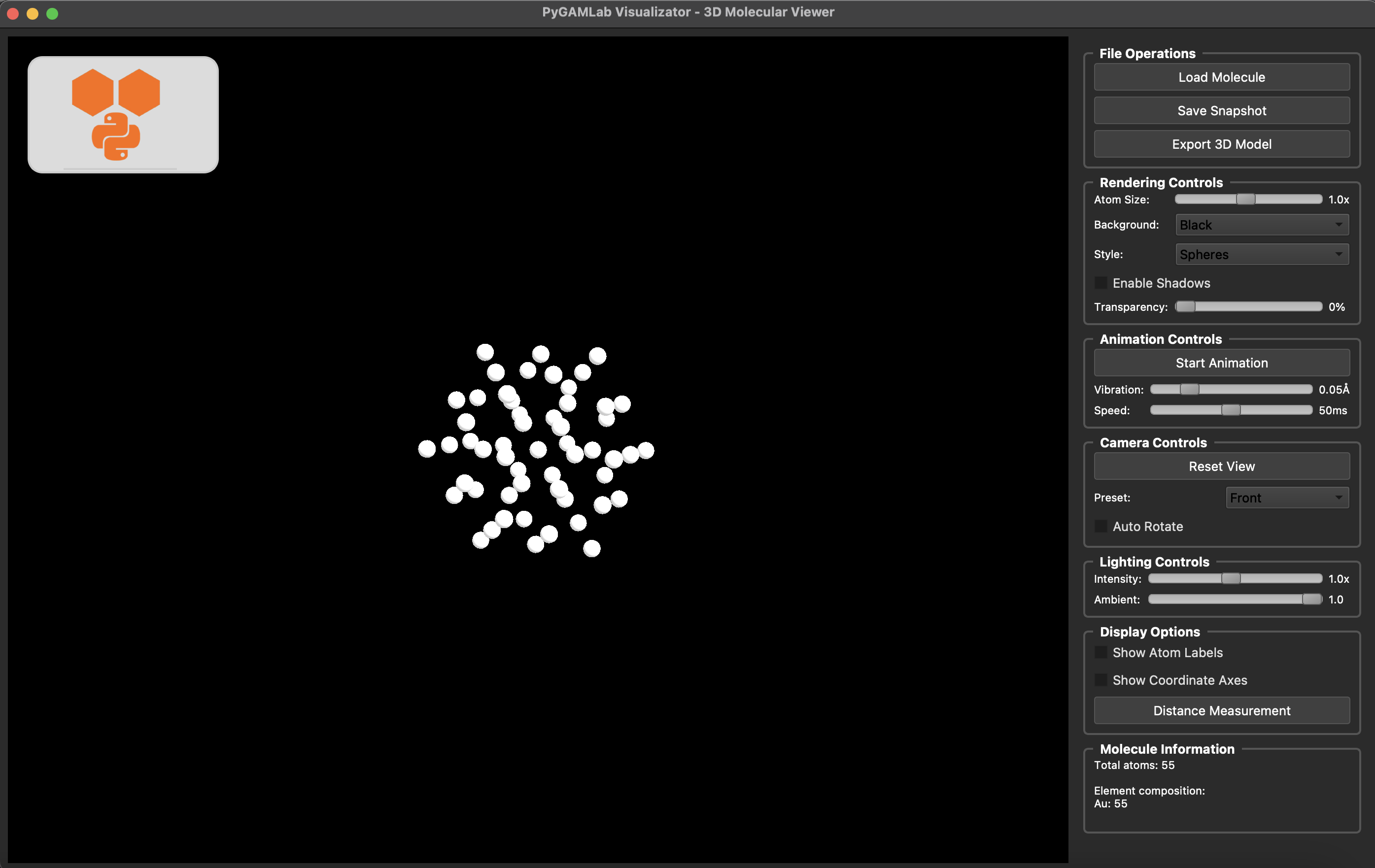
#also you can create One dimensional nanostructures like nanowires and nanotubes
from PyGamLab.structures.Generators import Nano_OneD_Builder
#You can create a carbon nanotube with length of 5.0 Angstrom and vacuum of 8.0 Angstrom
tube = Nano_OneD_Builder(material="C", structure_type="nanotube", length=5.0, vacuum=8.0)
#then you can use its methods like get_atoms to get the atoms object
tube_atoms = tube.get_atoms()
#for visulization you can use Molecular_Visulizer class
Molecular_Visualizer(tube_atoms, format='pyvista')
Visualizing 480 atoms using pyvista format...
/Users/apm/anaconda3/envs/DL/lib/python3.10/site-packages/pyvista/jupyter/notebook.py:56: UserWarning:
Failed to use notebook backend:
No module named 'trame'
Falling back to a static output.

#Like ZeroD and OneD nanostructures, you can create TwoD nanostructures like nanosheets and nanoribbons
from PyGamLab.structures.Generators import Nano_TwoD_Builder
builder_graphene = Nano_TwoD_Builder(material="graphene", structure_type="nanosheet")
graphene_atoms = builder_graphene.get_atoms()
Molecular_Visualizer(graphene_atoms, format='pyvista')
Visualizing 200 atoms using pyvista format...
/Users/apm/anaconda3/envs/DL/lib/python3.10/site-packages/pyvista/jupyter/notebook.py:56: UserWarning:
Failed to use notebook backend:
No module named 'trame'
Falling back to a static output.

#aLSO, you can create advanced alloys using AdvancedAlloys class
from PyGamLab.structures.Generators import AdvancedAlloys
alloy9 = AdvancedAlloys(
elements=["Au", "C"],
fractions=[0.6, 0.4],
metadata={"project": "test_alloy", "author": "Danial"}
)
alloy_atoms = alloy9.get_atoms()
Molecular_Visualizer(alloy_atoms, format='efficient_plotly')
Generated alloy with composition:
Au: 16 atoms (59.26%)
C: 11 atoms (40.74%)
Total atoms: 27
Crystal structure based on Au: fcc with lattice constant 4.08
Supercell size: (3, 3, 3)
Visualizing 27 atoms using efficient_plotly format...
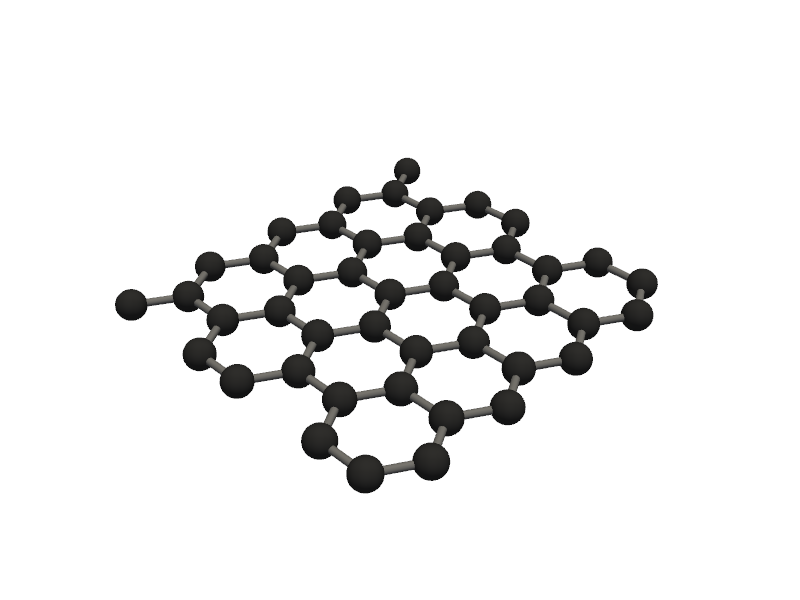
تاکنون همه چیزهایی که استفاده کردیم از ماژول Generator است که سطح بالای ASE با توابع پیشرفته است، همچنین پس از ایجاد هر اتم میتوانید توابع خاصی مانند translate، rotate و ... داشته باشید. از طرف دیگر ما GAM_architectures داریم که تمام کلاسهای مواد را از ابتدا میسازد و تاکنون در نسخه 2.0.0 شامل گرافن، سیلیسین، فسفورن، نانوذرات و نانولولهها میشود
#for instance you can directly use GAM_architectures to create specific materials like Graphene
from PyGamLab.structures.GAM_architectures import Graphene
builder = Graphene(width=10, length=10, edge_type='armchair')
graphene_atoms = builder.get_atoms()
Molecular_Visualizer(graphene_atoms, format='pyvista')
Visualizing 45 atoms using pyvista format...
/Users/apm/anaconda3/envs/DL/lib/python3.10/site-packages/pyvista/jupyter/notebook.py:56: UserWarning:
Failed to use notebook backend:
No module named 'trame'
Falling back to a static output.
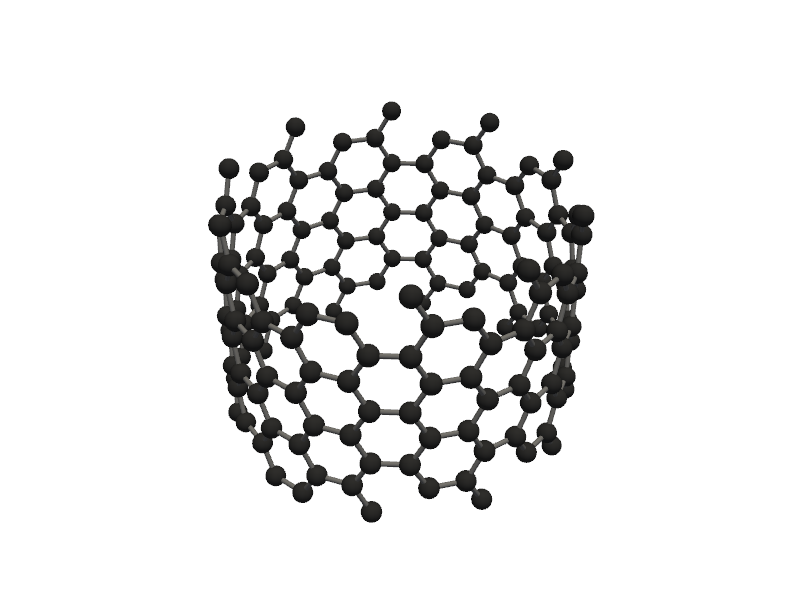
from PyGamLab.structures.GAM_architectures import Nanotube_Generator
nanotube = Nanotube_Generator(n=10, m=10, length=10.0, atom_type='C')
nanotube_atoms = nanotube.get_atoms()
Molecular_Visualizer(nanotube_atoms, format='pyvista')
Visualizing 160 atoms using pyvista format...
/Users/apm/anaconda3/envs/DL/lib/python3.10/site-packages/pyvista/jupyter/notebook.py:56: UserWarning:
Failed to use notebook backend:
No module named 'trame'
Falling back to a static output.
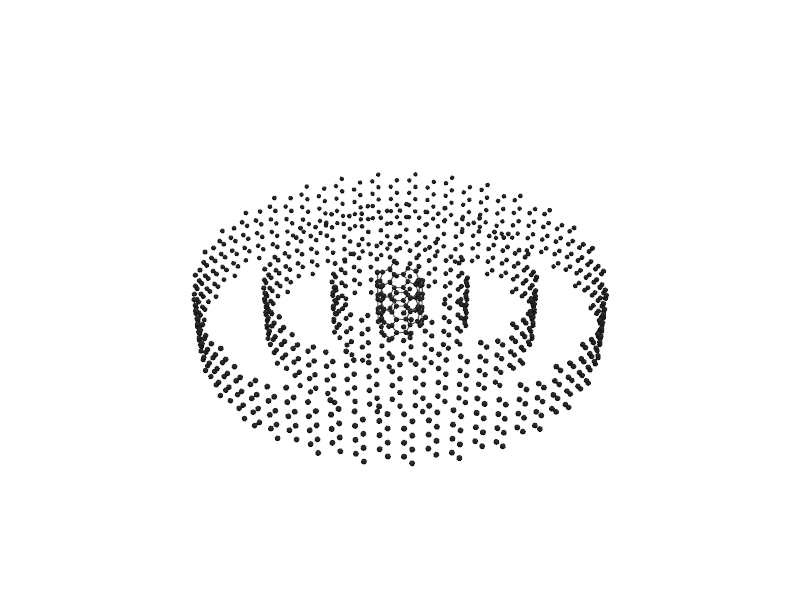
#lets go for more advanced nano tubes
nanotube = Nanotube_Generator(n=5, m=5, length=10.0, atom_type='C', multi_wall=[(10,10), (20, 20), (30,30)])
nanotube_atoms = nanotube.get_atoms()
Molecular_Visualizer(nanotube_atoms, format='pyvista')
Visualizing 1040 atoms using pyvista format...
/Users/apm/anaconda3/envs/DL/lib/python3.10/site-packages/pyvista/jupyter/notebook.py:56: UserWarning:
Failed to use notebook backend:
No module named 'trame'
Falling back to a static output.
#Also you can use Gamvis which is based on VTK for visualization
nanotube = Nanotube_Generator(n=5, m=5, length=30.0, atom_type='C', multi_wall=[(10,10), (20, 20), (30,30)])
nanotube_atoms = nanotube.get_atoms()
#just you need to change format to 'gamvis'
Molecular_Visualizer(nanotube_atoms, format='gamvis')
Visualizing 3120 atoms using gamvis format...
=== PyGAMLab Visualizator Debug Info ===
Python version: 3.10.13 | packaged by conda-forge | (main, Dec 23 2023, 15:35:25) [Clang 16.0.6 ]
PyQt5 version: 5.15.2
Platform: Darwin 24.3.0
Creating main window...
Loading 3120 atoms...
QPixmap::scaled: Pixmap is a null pixmap
qt.qpa.window: <QNSWindow: 0x3980feb20; contentView=<QNSView: 0x39559f020; QCocoaWindow(0x3955a5770, window=QWidgetWindow(0x395572f40, name="QWidgetClassWindow"))>> has active key-value observers (KVO)! These will stop working now that the window is recreated, and will result in exceptions when the observers are removed. Break in QCocoaWindow::recreateWindowIfNeeded to debug.
Showing window...
Initializing VTK interactor...
Application ready!
from PyGamLab.structures.GAM_architectures import Nanoparticle_Generator
npg = Nanoparticle_Generator(element="Au", size_nm=2.0)
npg_atoms = npg.get_atoms()
Molecular_Visualizer(npg_atoms, format='pyvista')
Visualizing 249 atoms using pyvista format...
/Users/apm/anaconda3/envs/DL/lib/python3.10/site-packages/pyvista/jupyter/notebook.py:56: UserWarning:
Failed to use notebook backend:
No module named 'trame'
Falling back to a static output.

مثالهای بیشتر در ….. موجود است
from PyGamLab.structures.GAM_architectures import Nanoparticle_Generator
npg = Nanoparticle_Generator(element="Au", size_nm=5.0, coating=("Cu", 2.0))
npg_atoms = npg.get_atoms()
Molecular_Visualizer(npg_atoms, format='gamvis')
Visualizing 7905 atoms using gamvis format...
=== PyGAMLab Visualizator Debug Info ===
Python version: 3.10.13 | packaged by conda-forge | (main, Dec 23 2023, 15:35:25) [Clang 16.0.6 ]
PyQt5 version: 5.15.2
Platform: Darwin 24.3.0
Creating main window...
Loading 7905 atoms...
QPixmap::scaled: Pixmap is a null pixmap
qt.qpa.window: <QNSWindow: 0x37e5ddd60; contentView=<QNSView: 0x3bdb09bd0; QCocoaWindow(0x37e5a3660, window=QWidgetWindow(0x37e5ae740, name="QWidgetClassWindow"))>> has active key-value observers (KVO)! These will stop working now that the window is recreated, and will result in exceptions when the observers are removed. Break in QCocoaWindow::recreateWindowIfNeeded to debug.
Showing window...
Initializing VTK interactor...
Application ready!
۵. پایگاههای داده¶
یکی از ضروریترین نیازهای محققان و مهندسان در علوم مواد دسترسی به پایگاههای داده قابل اعتماد است. چه در حال مطالعه ساختارهای بلوری، خواص مکانیکی، یا ویژگیهای الکترونیکی باشید، داشتن روشی یکپارچه برای پرسوجو و بازیابی داده زمان و تلاش زیادی را صرفهجویی میکند.
ماژول Databases PyGamLab برای سادهسازی این فرآیند به صورت یکپارچه و قابل دسترسی از یک پلتفرم واحد طراحی شده است.
چرا دسترسی به پایگاههای داده مهم است¶
محققان به دلایل مختلف به پایگاههای داده نیاز دارند:
طراحی مواد جدید با استفاده از دادههای مرجع (مانند پارامترهای شبکه، گروههای فضایی، یا ساختارهای باند الکترونیکی).
تأیید نتایج تجربی با مقایسه آنها با ادبیات گزارش شده یا دادههای شبیهسازی.
آموزش مدلهای هوش مصنوعی یا یادگیری ماشین برای پیشبینی خواص یا کشف مواد.
تولید نمونههای ساختار برای ورودیهای شبیهسازی.
انجام غربالگری با توان بالا برای مواد با خواص هدف (مثلاً باند گپ پایین، رسانایی بالا، و غیره).
databases PyGamLab این مشکل را با ارائه یک
API واحد و یکپارچه که با چندین پایگاه داده محبوب
ارتباط برقرار میکند حل میکند.پایگاههای داده پشتیبانی شده¶
در حال حاضر، ماژول Databases PyGamLab دسترسی به چندین پایگاه داده اصلی مواد را یکپارچه میکند:
- پایگاه داده باز بلورشناسی (COD) –مجموعهای منبع باز از ساختارهای بلوری، ایدهآل برای بازیابی مختصات اتمی، گروههای فضایی و پارامترهای سلول واحد برای ترکیبات معدنی و آلی.
- AFLOW (جریان خودکار برای کشف مواد) –یک پایگاه داده محاسباتی که خواص مکانیکی، حرارتی و الکترونیکی مواد را از محاسبات ab initio با توان بالا ارائه میدهد.
- Materials Project –یکی از پرکاربردترین پایگاههای داده مواد که ساختارهای باند، انرژیهای تشکیل، تانسورهای الاستیک و اطلاعات تقارن را برای هزاران ترکیب ارائه میدهد.
- JARVIS (NIST) –یک پایگاه داده جامع مواد که توسط NIST توسعه یافته و شامل دادههای مکانیکی کوانتومی، یادگیری ماشین و دادههای تجربی در طیف گستردهای از مواد است.
چالش استفاده از چندین پایگاه داده¶
هر یک از این پایگاههای داده با روش دسترسی خاص خود همراه است:
برخی فقط از طریق رابطهای گرافیکی مبتنی بر وب (GUIs) در دسترس هستند که کاربران به صورت دستی مواد را جستجو میکنند و داده دانلود میکنند.
برخی دیگر پوششهای پایتون ارائه میدهند که نیاز به نصب جداگانه و سینتکس پرسوجوی متفاوت دارند.
بسیاری از APIهای RESTful پشتیبانی میکنند که نیاز به ساخت درخواستهای HTTP، مدیریت endpointها و مدیریت پاسخهای JSON دارند — اغلب با ساختارهای کمی متفاوت.
این تنوع باعث میشود که ترکیب داده از پایگاههای داده مختلف یا انجام تحلیلهای خودکار و در مقیاس بزرگ دشوار باشد.
راهحل یکپارچه PyGamLab: Explorer¶
برای حل این مشکل، PyGamLab کلاس ``Explorer`` را در ماژول Databases معرفی میکند — یک رابط یکپارچه که شما را به چندین منبع داده با استفاده از سینتکس و منطق سازگار متصل میکند.
Explorer میتوانید: - داده را از چندین منبع بازیابی کنید
(COD، AFLOW، Materials Project، JARVIS).نیازی به نگرانی در مورد endpointها، tokenها یا فرمتهای ناسازگار ندارید — PyGamLab ارتباطات را مدیریت میکند و اشیاء داده تمیز و استاندارد را برمیگرداند که میتوانند مستقیماً در گردش کار شما استفاده شوند.
مزایای ماژول Databases¶
دسترسی یکپارچه – یک رابط سازگار برای چندین پایگاه داده.
همگامسازی بین پایگاههای داده – بازیابی و مقایسه داده از چندین منبع به صورت همزمان.
آماده خودکارسازی – ادغام مستقیم در خطوط لوله داده یا گردش کارهای هوش مصنوعی شما.
خروجی استاندارد – دیگر فرمتها یا سردرگمی API ناسازگار وجود ندارد.
قابل گسترش – پایگاههای داده جدید را میتوان به راحتی در نسخههای آینده اضافه کرد.
مثال استفاده¶
#میتوانید داده را از AFLOW دریافت کنید
from PyGamLab.databases import Aflow_Explorer
my_explorer=Aflow_Explorer()
my_explorer.search_materials(formula="Cs1F3Mg1", max_results=3,batch_size=10)
[{'auid': 'aflow:141bd22d5b219f1f',
'prototype': 'Cs1F3Mg1_ICSD_290359',
'spacegroup_relax': 221,
'dft_type': ['PAW_PBE'],
'spinD': array([0, 0, 0, 0, 0]),
'spinF': 0,
'enthalpy_formation_cell': -15.9531,
'natoms': 5},
{'auid': 'aflow:494166da0e7a6134',
'prototype': 'Cs1F3Mg1_ICSD_49584',
'spacegroup_relax': 221,
'dft_type': ['PAW_PBE'],
'spinD': array([0, 0, 0, 0, 0]),
'spinF': 0,
'enthalpy_formation_cell': -15.9536,
'natoms': 5},
{'auid': 'aflow:4c5ca27e65d9772c',
'prototype': 'T0009.CAB',
'spacegroup_relax': 221,
'dft_type': ['PAW_PBE'],
'spinD': array([0, 0, 0, 0, 0]),
'spinF': 0,
'enthalpy_formation_cell': -6.56372,
'natoms': 5}]
#اکنون شما شناسههای مختلف aflow دارید و همچنین میتوانید مشخص کنید کدام یک را میخواهید تا خواص آنها را دریافت کنید
#میتوانید خواص الکتریکی، مکانیکی و .... دریافت کنید
#مثلاً auid 'aflow:141bd22d5b219f1f' مناسب است
electronic_data=my_explorer.fetch_electronic_properties(auid='aflow:141bd22d5b219f1f')
print(electronic_data)
{'formula': 'Cs1F3Mg1', 'bandgap': 6.721, 'bandgap_fit': 9.97291, 'bandgap_type': 'insulator-direct', 'delta_elec_energy_convergence': 1.2486e-05, 'delta_elec_energy_threshold': 0.0001, 'ldau_TLUJ': None, 'dft_type': ['PAW_PBE'], 'bader_atomic_volumes': array([27.44 , 14.2738, 14.2738, 14.2745, 6.4306]), 'bader_net_charges': array([ 0.9138, -0.8733, -0.8733, -0.8734, 1.7062]), 'spinD': array([0, 0, 0, 0, 0]), 'spinF': 0, 'spin_atom': 0, 'spin_cell': 0, 'scintillation_attenuation_length': 2.4419}
#همچنین میتوانید خواص مکانیکی را دریافت کنید
mechanical_data=my_explorer.fetch_mechanical_properties(auid='aflow:141bd22d5b219f1f')
print(mechanical_data)
{'formula': 'Cs1F3Mg1', 'bulk_modulus_reuss': None, 'bulk_modulus_voigt': None, 'bulk_modulus_vrh': None, 'shear_modulus_reuss': None, 'shear_modulus_voigt': None, 'shear_modulus_vrh': None, 'poisson_ratio': None, 'elastic_anisotropy': None, 'stress_tensor': array([ 0.97, 0. , -0. , 0. , 0.97, 0. , -0. , 0. , 0.97]), 'forces': array([[ 0., 0., 0.],
[-0., 0., 0.],
[ 0., 0., 0.],
[ 0., 0., -0.],
[ 0., 0., 0.]]), 'Pulay_stress': 0, 'pressure': 0, 'pressure_residual': 0.97}
#همچنین میتوانید خواص ترمودینامیکی را دریافت کنید
thermodynamic_data=my_explorer.fetch_thermodynamic_properties(auid='aflow:141bd22d5b219f1f')
print(thermodynamic_data)
{'formula': 'Cs1F3Mg1', 'acoustic_debye': None, 'debye': None, 'gruneisen': None, 'heat_capacity_Cp_300K': None, 'heat_capacity_Cv_300K': None, 'thermal_conductivity_300K': None, 'thermal_expansion_300K': None, 'bulk_modulus_isothermal_300K': None, 'bulk_modulus_static_300K': None, 'entropy_atom': 0.00118258, 'entropy_cell': 0.00591288, 'enthalpy_atom': -4.79552, 'enthalpy_cell': -23.9776, 'enthalpy_formation_atom': -3.19062, 'enthalpy_formation_cell': -15.9531, 'energy_atom': -4.79552, 'energy_cell': -23.9776, 'energy_cutoff': array([560]), 'entropic_temperature': 38963.2}
#همچنین میتوانید ساختار را دریافت کنید
structure_data=my_explorer.fetch_structure(auid='aflow:141bd22d5b219f1f')
print(structure_data)
{'formula': 'Cs1F3Mg1', 'Bravais_lattice_orig': 'CUB', 'Bravais_lattice_relax': 'CUB', 'Pearson_symbol_orig': 'cP5', 'Pearson_symbol_relax': 'cP5', 'lattice_system_orig': 'cubic', 'lattice_system_relax': 'cubic', 'lattice_variation_orig': 'CUB', 'lattice_variation_relax': 'CUB', 'spacegroup_orig': 221, 'spacegroup_relax': 221, 'sg': ['Pm-3m #221', 'Pm-3m #221', 'Pm-3m #221'], 'sg2': ['Pm-3m #221', 'Pm-3m #221', 'Pm-3m #221'], 'prototype': 'Cs1F3Mg1_ICSD_290359', 'stoich': [0.2, 0.6, 0.2], 'stoichiometry': array([0.2, 0.6, 0.2]), 'geometry': array([ 4.2486519, 4.2486519, 4.2486519, 90. , 90. ,
90. ]), 'natoms': 5, 'nspecies': 3, 'nbondxx': array([4.2487, 3.0043, 3.6794, 3.0043, 2.1243, 4.2487]), 'composition': array([1, 3, 1]), 'compound': 'Cs1F3Mg1', 'species': ['Cs', 'F', 'Mg'], 'species_pp': ['Cs_sv', 'F', 'Mg_pv'], 'species_pp_ZVAL': array([9, 7, 8]), 'species_pp_version': ['Cs_sv:PAW_PBE:08Apr2002', 'F:PAW_PBE:08Apr2002', 'Mg_pv:PAW_PBE:06Sep2000'], 'positions_cartesian': array([[0. , 0. , 0. ],
[2.12433, 2.12433, 0. ],
[0. , 2.12433, 2.12433],
[2.12433, 0. , 2.12433],
[2.12433, 2.12433, 2.12433]]), 'positions_fractional': array([[0. , 0. , 0. ],
[0.5, 0.5, 0. ],
[0. , 0.5, 0.5],
[0.5, 0. , 0.5],
[0.5, 0.5, 0.5]]), 'valence_cell_iupac': 6, 'valence_cell_std': 24, 'volume_atom': 15.3385, 'volume_cell': 76.6926, 'density': 4.63796}
#همچنین برای کارایی بیشتر میتوانید تمام خواص را یکجا دریافت کنید
all_data=my_explorer.fetch_all_data(auid='aflow:141bd22d5b219f1f')
print(all_data)
{'electronic_prop': {'formula': 'Cs1F3Mg1', 'bandgap': 6.721, 'bandgap_fit': 9.97291, 'bandgap_type': 'insulator-direct', 'delta_elec_energy_convergence': 1.2486e-05, 'delta_elec_energy_threshold': 0.0001, 'ldau_TLUJ': None, 'dft_type': ['PAW_PBE'], 'bader_atomic_volumes': array([27.44 , 14.2738, 14.2738, 14.2745, 6.4306]), 'bader_net_charges': array([ 0.9138, -0.8733, -0.8733, -0.8734, 1.7062]), 'spinD': array([0, 0, 0, 0, 0]), 'spinF': 0, 'spin_atom': 0, 'spin_cell': 0, 'scintillation_attenuation_length': 2.4419}, 'mechanical_prop': {'formula': 'Cs1F3Mg1', 'bulk_modulus_reuss': None, 'bulk_modulus_voigt': None, 'bulk_modulus_vrh': None, 'shear_modulus_reuss': None, 'shear_modulus_voigt': None, 'shear_modulus_vrh': None, 'poisson_ratio': None, 'elastic_anisotropy': None, 'stress_tensor': array([ 0.97, 0. , -0. , 0. , 0.97, 0. , -0. , 0. , 0.97]), 'forces': array([[ 0., 0., 0.],
[-0., 0., 0.],
[ 0., 0., 0.],
[ 0., 0., -0.],
[ 0., 0., 0.]]), 'Pulay_stress': 0, 'pressure': 0, 'pressure_residual': 0.97}, 'thermo_prop': {'formula': 'Cs1F3Mg1', 'acoustic_debye': None, 'debye': None, 'gruneisen': None, 'heat_capacity_Cp_300K': None, 'heat_capacity_Cv_300K': None, 'thermal_conductivity_300K': None, 'thermal_expansion_300K': None, 'bulk_modulus_isothermal_300K': None, 'bulk_modulus_static_300K': None, 'entropy_atom': 0.00118258, 'entropy_cell': 0.00591288, 'enthalpy_atom': -4.79552, 'enthalpy_cell': -23.9776, 'enthalpy_formation_atom': -3.19062, 'enthalpy_formation_cell': -15.9531, 'energy_atom': -4.79552, 'energy_cell': -23.9776, 'energy_cutoff': array([560]), 'entropic_temperature': 38963.2}, 'structure': {'formula': 'Cs1F3Mg1', 'Bravais_lattice_orig': 'CUB', 'Bravais_lattice_relax': 'CUB', 'Pearson_symbol_orig': 'cP5', 'Pearson_symbol_relax': 'cP5', 'lattice_system_orig': 'cubic', 'lattice_system_relax': 'cubic', 'lattice_variation_orig': 'CUB', 'lattice_variation_relax': 'CUB', 'spacegroup_orig': 221, 'spacegroup_relax': 221, 'sg': ['Pm-3m #221', 'Pm-3m #221', 'Pm-3m #221'], 'sg2': ['Pm-3m #221', 'Pm-3m #221', 'Pm-3m #221'], 'prototype': 'Cs1F3Mg1_ICSD_290359', 'stoich': [0.2, 0.6, 0.2], 'stoichiometry': array([0.2, 0.6, 0.2]), 'geometry': array([ 4.2486519, 4.2486519, 4.2486519, 90. , 90. ,
90. ]), 'natoms': 5, 'nspecies': 3, 'nbondxx': array([4.2487, 3.0043, 3.6794, 3.0043, 2.1243, 4.2487]), 'composition': array([1, 3, 1]), 'compound': 'Cs1F3Mg1', 'species': ['Cs', 'F', 'Mg'], 'species_pp': ['Cs_sv', 'F', 'Mg_pv'], 'species_pp_ZVAL': array([9, 7, 8]), 'species_pp_version': ['Cs_sv:PAW_PBE:08Apr2002', 'F:PAW_PBE:08Apr2002', 'Mg_pv:PAW_PBE:06Sep2000'], 'positions_cartesian': array([[0. , 0. , 0. ],
[2.12433, 2.12433, 0. ],
[0. , 2.12433, 2.12433],
[2.12433, 0. , 2.12433],
[2.12433, 2.12433, 2.12433]]), 'positions_fractional': array([[0. , 0. , 0. ],
[0.5, 0.5, 0. ],
[0. , 0.5, 0.5],
[0.5, 0. , 0.5],
[0.5, 0.5, 0.5]]), 'valence_cell_iupac': 6, 'valence_cell_std': 24, 'volume_atom': 15.3385, 'volume_cell': 76.6926, 'density': 4.63796}, 'meta_data': {}}
#بنابراین میتوانید از سایر پایگاههای داده مانند Materials Project، Jarvis، COD نیز استفاده کنید
#فقط میتوانید explorer مختلف ایجاد کنید اما متدها مشابه هستند
#برای COD -->
from PyGamLab.databases import COD_Explorer
my_explorer_cod=COD_Explorer()
#برای Jarvis -->
from PyGamLab.databases import Jarvis_Explorer
my_jarvis_explorer=Jarvis_Explorer()
#برای Materials Project -->
from PyGamLab.databases import MaterialsProject_Explorer
#فقط توجه داشته باشید که برای Materials Project باید یک کلید API از وبسایت آنها داشته باشید
#میتوانید ثبتنام کنید و کلید API خود را دریافت کرده و اینجا قرار دهید
#وبسایت: https://next-gen.materialsproject.org/api
my_explorer_mp=MaterialsProject_Explorer(api_key="your_api_key_here")
#سپس روی شیء خود میتوانید از متدهای مشابه مانند search_materials، fetch_electronic_properties و ..... استفاده کنید
همچنین Gamlab روش کارآمدتری برای دریافت داده از پایگاههای داده ارائه میدهد. میتوانید از GAM_Explorer استفاده کنید که میتواند به چندین پایگاه داده به صورت همزمان متصل شود و یک wrapper برای explorerهای مختلف است و میتوانید آنها را با مشخص کردن backend فراخوانی کنید
from PyGamLab.databases import GAM_Explorer
gam_explorer=GAM_Explorer(backend='backend شما اینجا مانند aflow، jarvis، mp، cod')
میتوانید backend را در آنجا مشخص کنید و برای مثال backend='aflow' یا backend='jarvis' یا backend='mp' یا backend='cod' و به ترتیب به Aflow_Explorer، Jarvis_Explorer، MaterialsProject_Explorer، COD_Explorer تبدیل میشود و سپس میتوانید از متدهای مشابه مانند search_materials، fetch_electronic_properties و ... استفاده کنید
و سپس میتوانید ببینید داده از کدام پایگاه داده دریافت شده است
۶. تحلیل دادههای نانو¶
برای سادهسازی این فرآیند، PyGamLab ماژول ``data_analysis`` را معرفی میکند — مجموعه قدرتمندی از ابزارها که برای کمک به محققان برای بارگذاری، تحلیل و نمایش دادههایشان با حداقل تلاش طراحی شده است.
چرا ماژول تحلیل داده PyGamLab؟¶
در علوم مواد تجربی و محاسباتی، محققان اغلب با چالشهایی مانند موارد زیر روبرو میشوند:
نرمافزارهای ابزار فقط تصاویر به جای دادههای عددی خروجی میدهند.
فرمتهای داده ناسازگار (CSV، TXT، Excel، و غیره).
نیاز به توابع تحلیل سفارشی برای هر نوع آزمایش.
مراحل رسم دستی و پاکسازی داده که برای هر مجموعه داده تکرار میشود.
فلسفه طراحی اصلی¶
data_analysis برای
مطابقت با یک تکنیک تجربی خاص یا ابزار
ویژگییابی طراحی شده است.فلسفه ساده است: > شما روی علم خود تمرکز کنید و PyGamLab مراقب منطق مدیریت و تحلیل داده است.
نحوه عملکرد¶
- آمادهسازی دادههای خوددادههای شما باید در فرمت ``pandas.DataFrame`` باشند که در آن هر ستون یک متغیر اندازهگیری شده را نشان میدهد (مثلاً: طول موج، شدت، زمان، ولتاژ، و غیره).
- انتخاب تابع تحلیل خودهر روش تجربی (مثلاً XRD، Raman، IV، TEM، و غیره) دارای تابع تخصصی خود در ماژول
data_analysisاست. - مشخص کردن نوع کاربرداکثر توابع یک آرگومان به نام
applicationمیپذیرند که تعیین میکند چه عملی میخواهید انجام دهید.گزینههای رایج شامل:"plot"→ نمایش دادههای خود در سبکهای مختلف (خطی، پراکندگی، مقیاس لگاریتمی، و غیره)"calculate"→ محاسبه نتایج کمی (مانند موقعیتهای پیک، نسبتهای شدت، و غیره)"process"→ انجام حذف پسزمینه، هموارسازی، نرمالسازی، یا پردازش پیشرفته سیگنال
- اجرا و تفسیر نتایجخروجی میتواند یک نمودار، داده پردازش شده، یا نتایج عددی محاسبه شده آماده برای انتشار یا محاسبه بیشتر باشد.
مثال: تحلیل دادههای NMR¶
#ابتدا باید pandas را import کنید
import pandas as pd
#pd دو تابع دارد یکی read_csv برای خواندن فایلهای csv و دیگری read_excel برای خواندن فایلهای excel
#برای خواندن فایلهای csv
data_csv=pd.read_csv('your_file_name.csv')
#برای خواندن فایلهای excel
data_excel=pd.read_excel('your_file_name.xlsx')
from PyGamLab.Data_Analysis import NMR_Analysis
#این تابع داده را به صورت dataframe و پارامتر application دریافت میکند
#پارامتر application به معنای نوع تحلیلی است که میخواهید انجام دهید
#مثلاً رسم نمودار
NMR_Analysis(data=data_csv, application='plot')
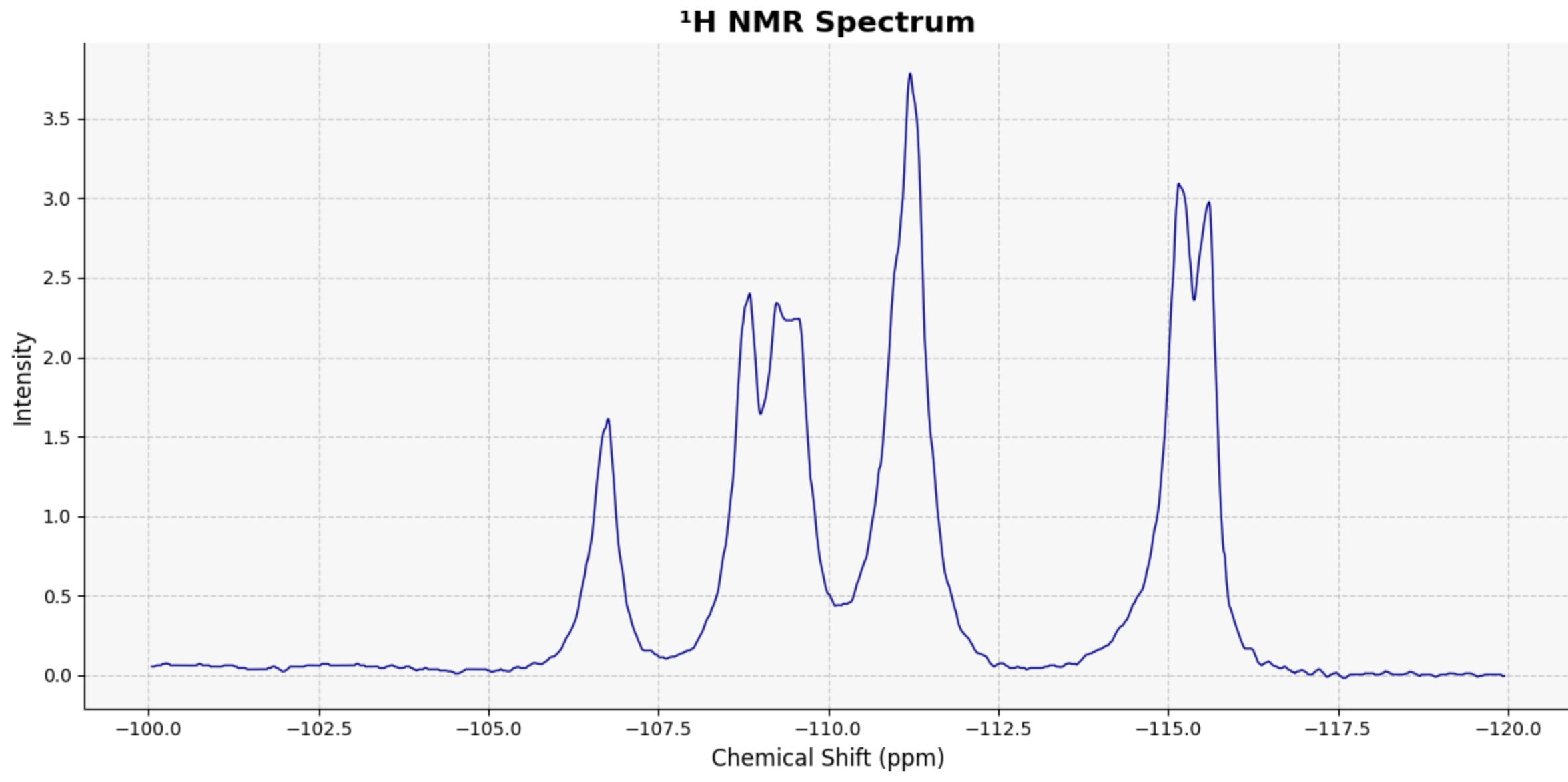
my_peak_regions = {
'Aromatic': (-107, -105),
'Benzylic CH₂': (-112, -108),
'Acetyl CH₃': (-118, -113)
}
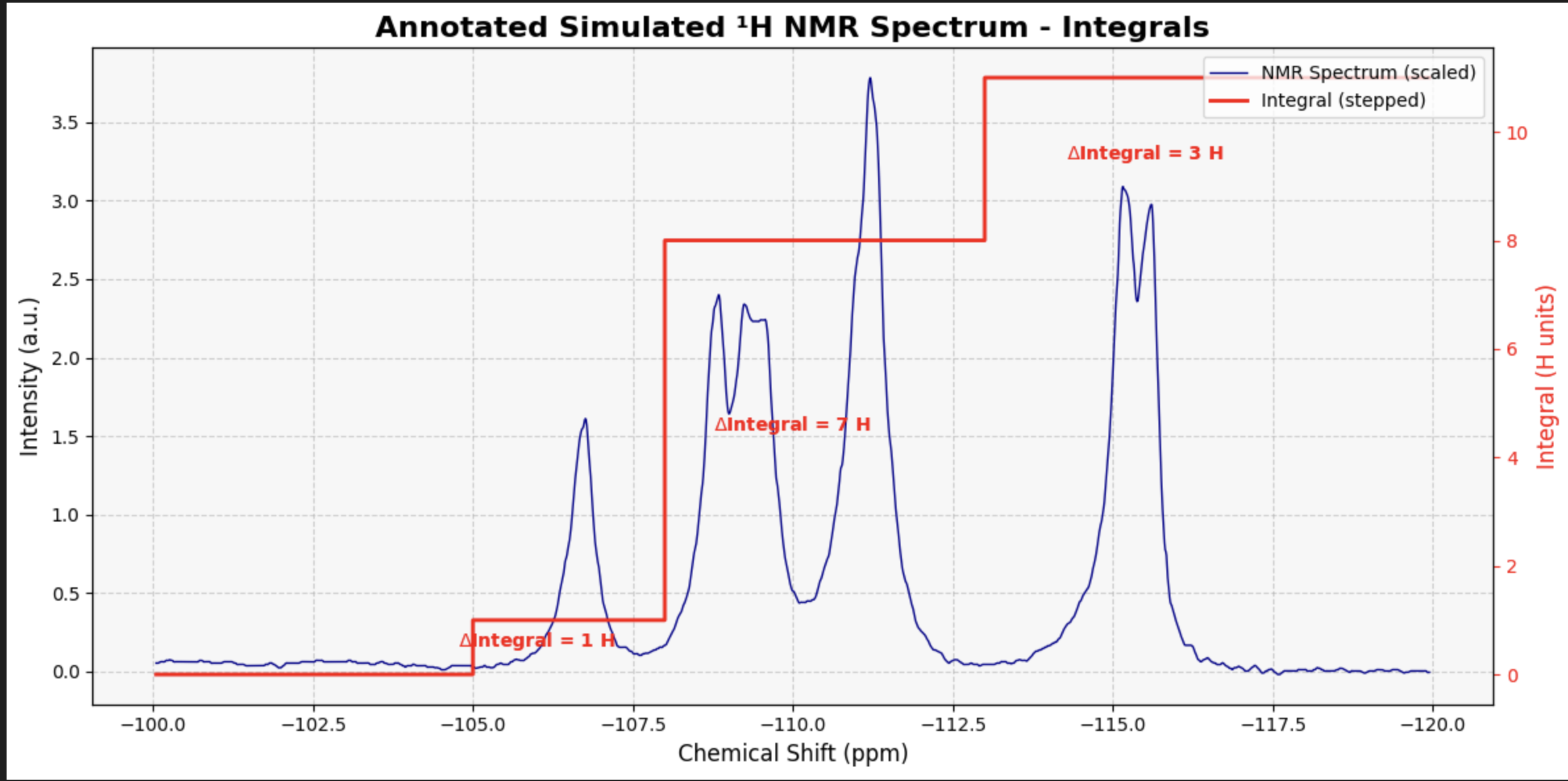
# فراخوانی تابع برای تولید نمودار با مراحل انتگرال
NMR_Analysis(data, application='plot_with_integrals', peak_regions=my_peak_regions)
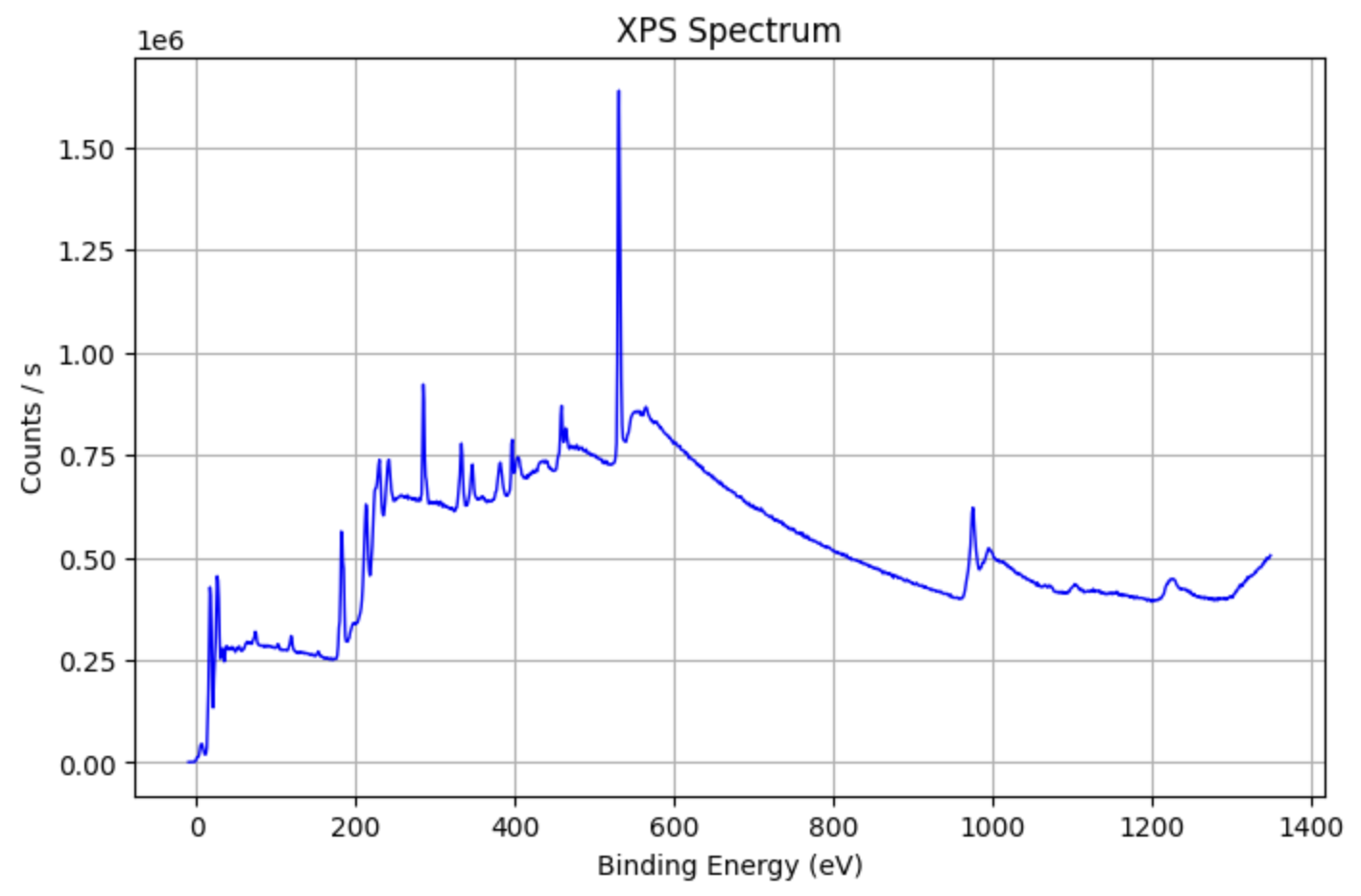
مثال: تحلیل دادههای XPS¶
XPS_data=pd.read_excel('your_file_name.xlsx')
from PyGamLab.Data_Analysis import XPS_Analysis
XPS_Analysis(data=XPS_data, application='plot')
peaks = XPS_Analysis(df, application='peak_detection', peak_prominence=10000)
for p in peaks:
print(f"Peak at {p['energy']:.2f} eV, height {p['counts']:.1f}, FWHM {p['width']:.2f} points")
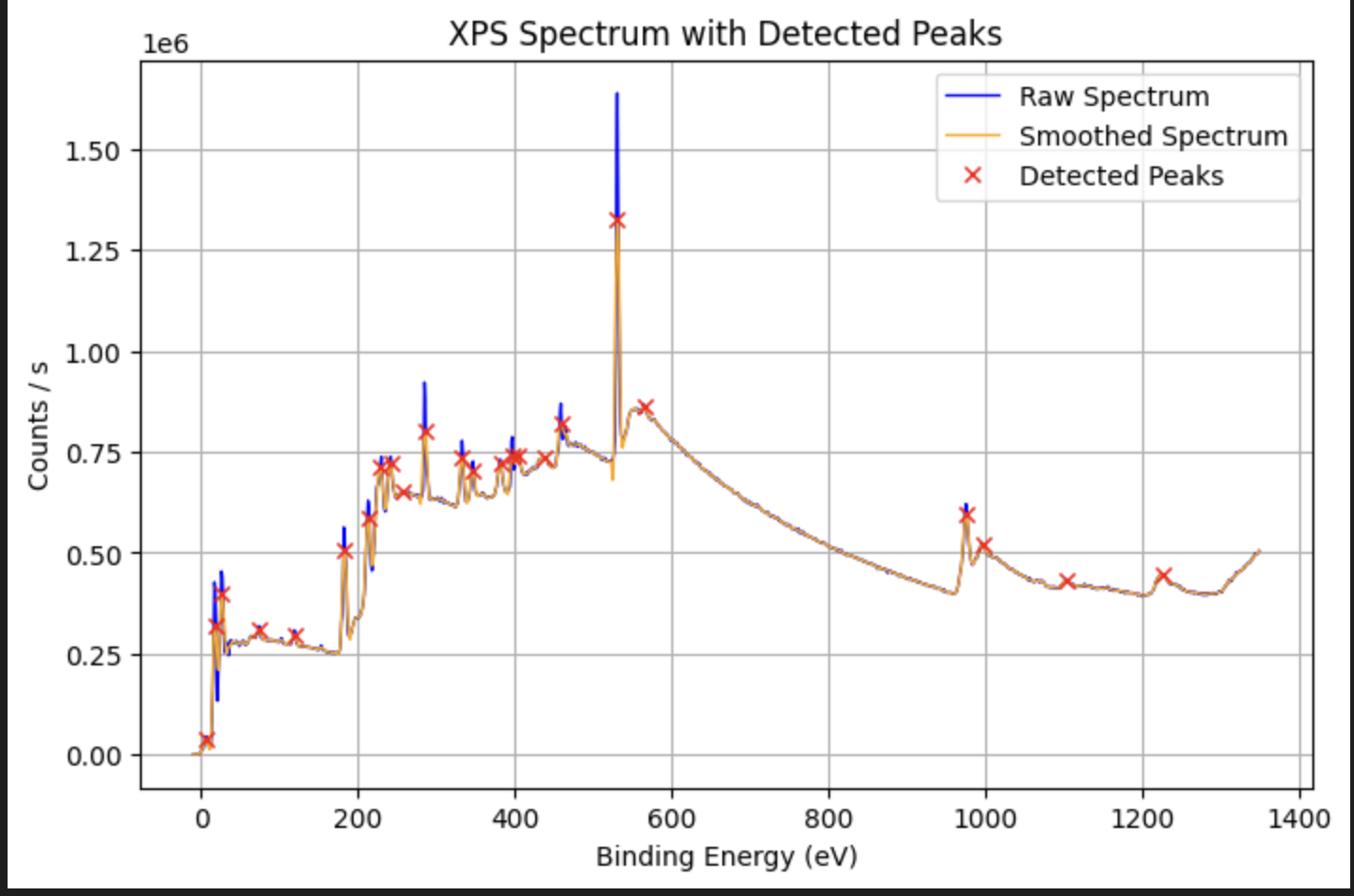
#Peak at 1227.08 eV, height 447819.0, FWHM 23.11 points
#Peak at 1105.08 eV, height 433361.0, FWHM 10.59 points
#Peak at 997.08 eV, height 517901.0, FWHM 13.04 points
#Peak at 976.08 eV, height 621396.0, FWHM 9.79 points
#Peak at 565.08 eV, height 867297.0, FWHM 42.23 points
#Peak at 531.08 eV, height 1638660.0, FWHM 8.20 points
#Peak at 460.08 eV, height 815931.0, FWHM 42.35 points
#Peak at 438.08 eV, height 731991.0, FWHM 13.62 points
#Peak at 404.08 eV, height 745177.0, FWHM 12.58 points
#Peak at 398.08 eV, height 728148.0, FWHM 3.80 points
#Peak at 382.08 eV, height 731348.0, FWHM 7.37 points
#Peak at 347.08 eV, height 726994.0, FWHM 6.32 points
#Peak at 333.08 eV, height 778125.0, FWHM 6.33 points
#Peak at 286.08 eV, height 879292.0, FWHM 6.50 points
#Peak at 257.08 eV, height 651998.0, FWHM 16.93 points
#Peak at 242.08 eV, height 738299.0, FWHM 6.83 points
#Peak at 229.08 eV, height 725264.0, FWHM 7.39 points
#Peak at 213.08 eV, height 629412.0, FWHM 5.06 points
#Peak at 183.08 eV, height 559701.0, FWHM 6.10 points
#Peak at 119.08 eV, height 308560.0, FWHM 5.81 points
#Peak at 74.08 eV, height 319002.0, FWHM 37.19 points
#Peak at 26.08 eV, height 454384.0, FWHM 5.36 points
#Peak at 18.08 eV, height 406393.0, FWHM 4.21 points
#Peak at 7.08 eV, height 45308.4, FWHM 5.49 points
DSC¶
from PyGamLab.Data_Analysis import DSC
dsc_data=pd.read_excel('your_file_name.xlsx')
DSC(data=dsc_data, application='plot')
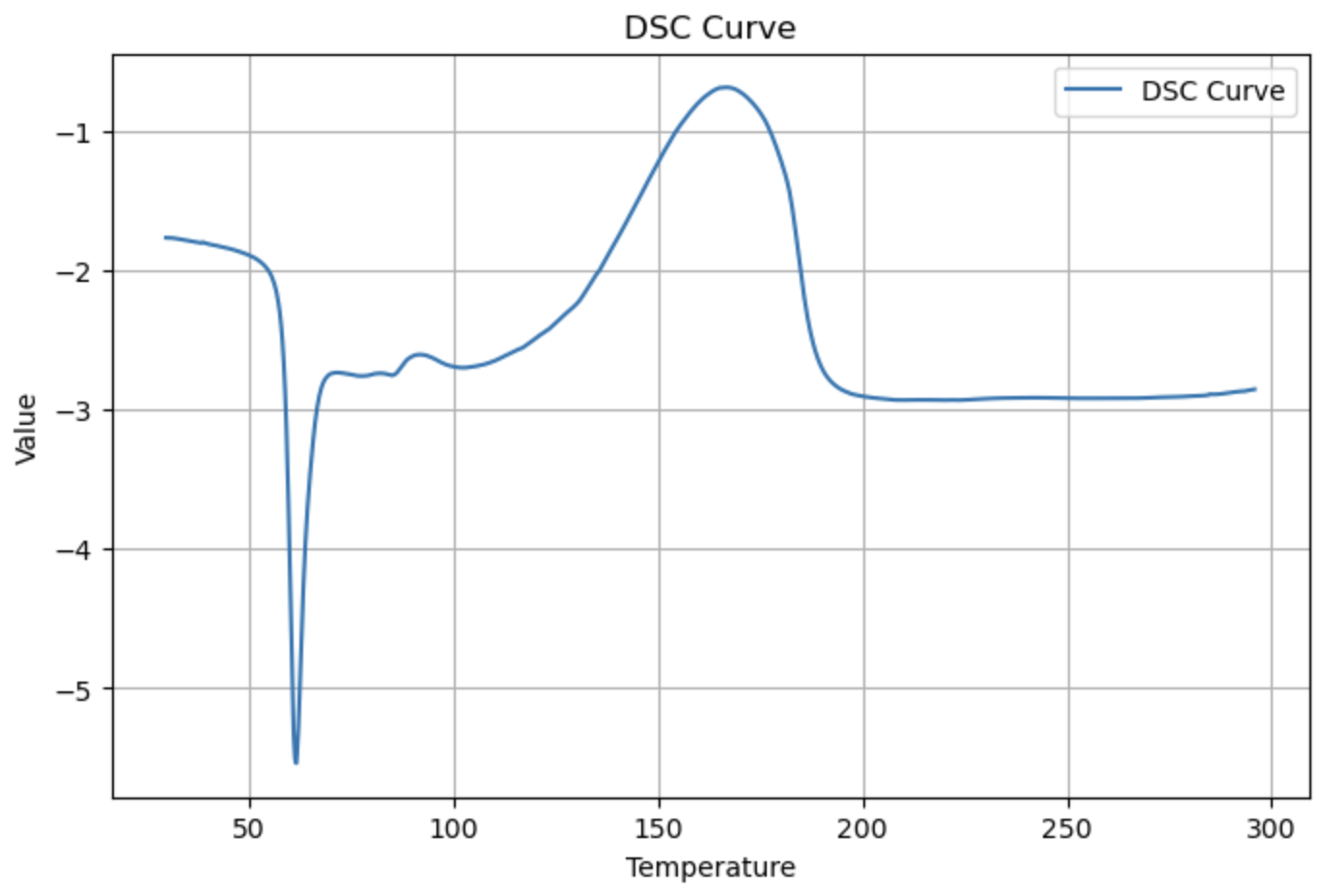
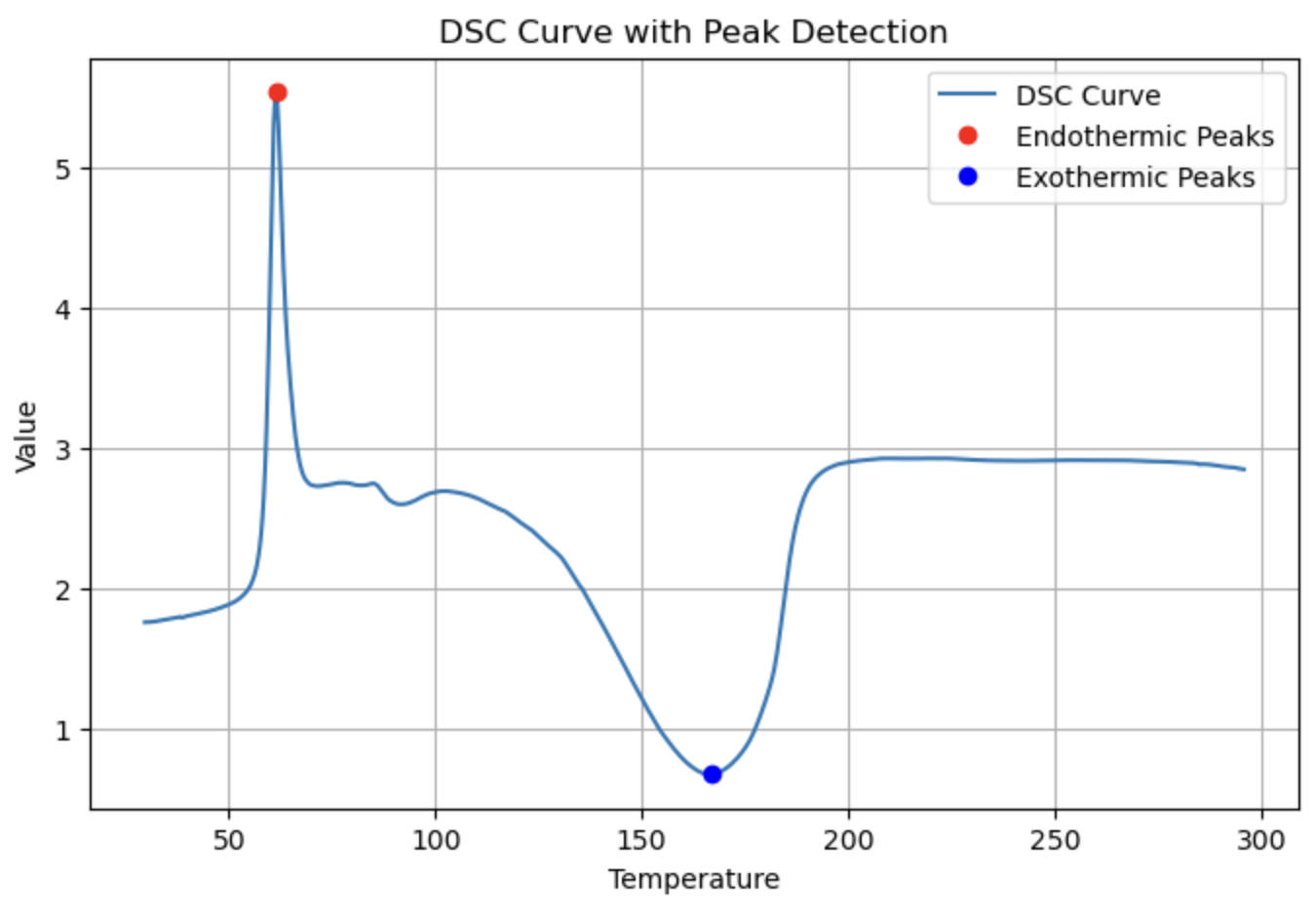
peaks = DSC(dsc_data, application="peak_detection", prominence=2.0, distance=10)
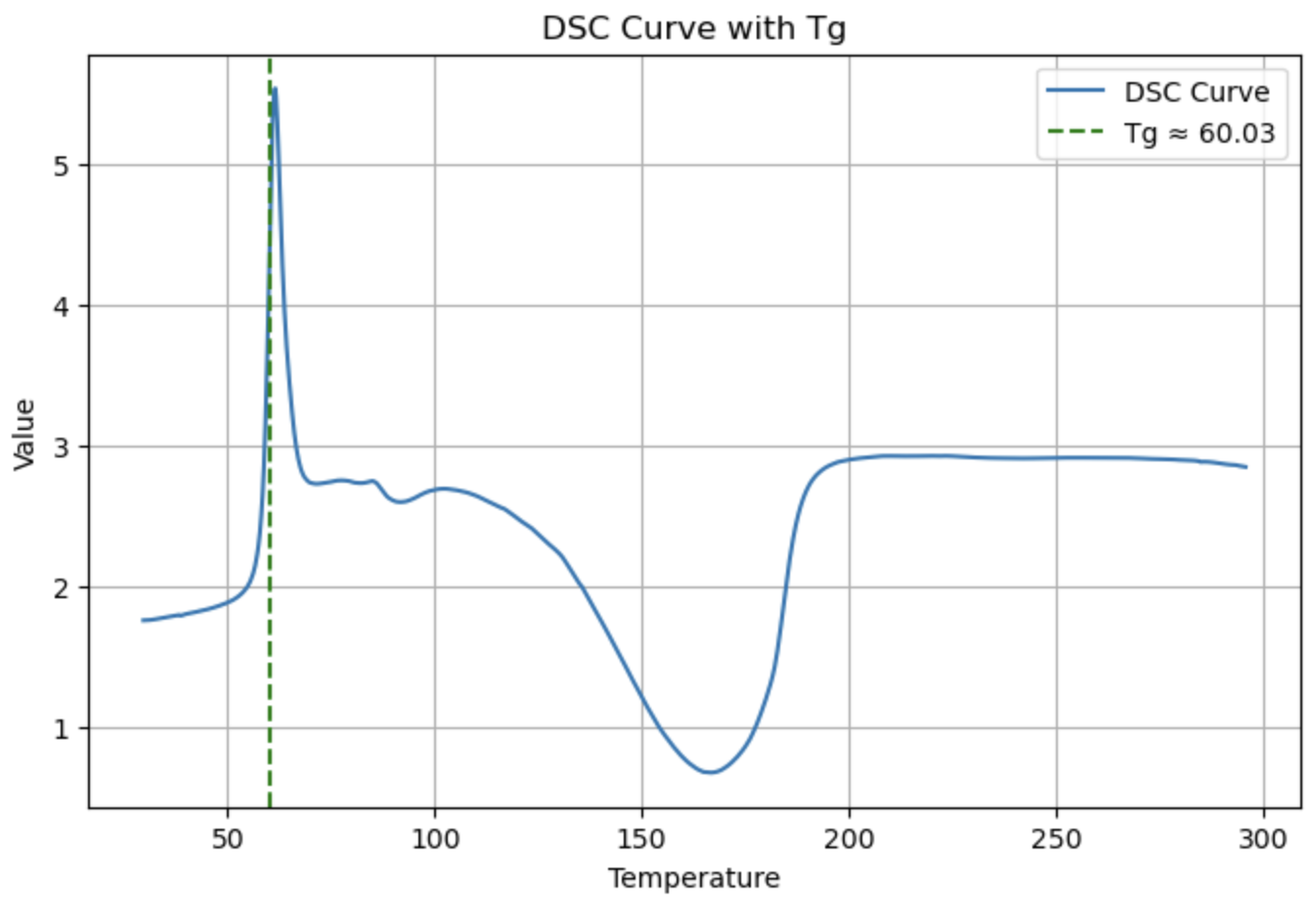
DSC(dsc_data, application="Tg")
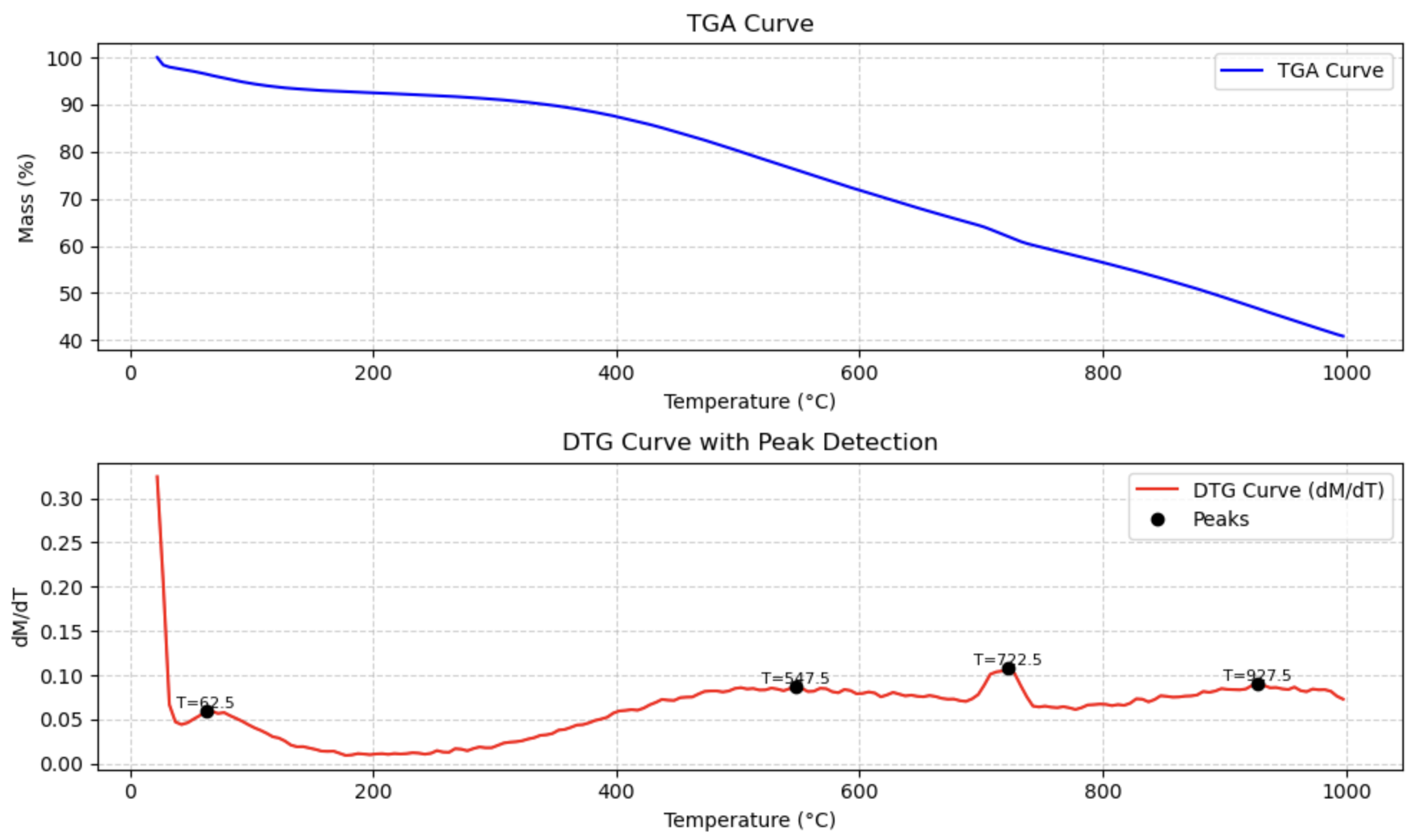
TGA¶
from PyGamLab.Data_Analysis import TGA
data=pd.read_excel('your_file_name.xlsx')
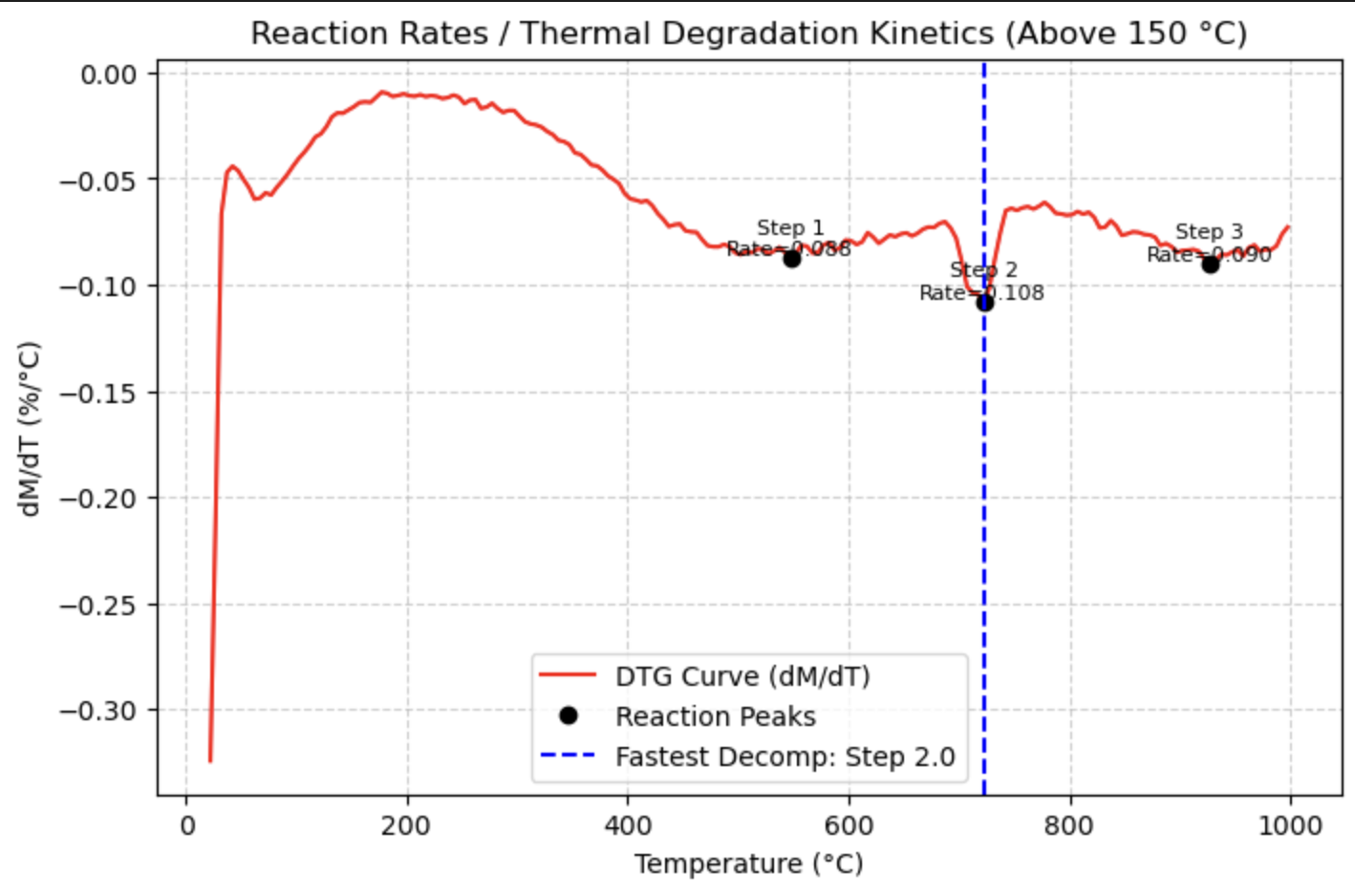
TGA(data, application="peaks")
TGA(data, application="kinetics")
۷. هسته هوش مصنوعی — مدلهای هوشمند برای علوم مواد¶
🌍 نمای کلی¶
در عصر هوش مصنوعی، پیشرفتها در علوم و مهندسی به طور فزایندهای توسط مدلهای یادگیری ماشین که میتوانند از دادهها یاد بگیرند، الگوهای پیچیده را تعمیم دهند و پیشبینی کنند سریعتر و دقیقتر از روشهای سنتی هدایت میشوند.
پس از انقلاب معماریهای ترنسفورمر و تکنیکهای تنظیم دقیق، سیستمهای هوش مصنوعی مدرن — از مدلهای زبان بزرگ (LLM) مانند GPT تا پیشبینیکنندههای خاص دامنه — نحوه پردازش اطلاعات را دگرگون کردهاند. با این حال، در حالی که زمینههایی مانند پردازش زبان طبیعی و بینایی کامپیوتر دسترسی گسترده به مدلهای از پیش آموزش دیده و تنظیم شده از طریق پلتفرمهایی مانند Hugging Face دارند، علوم مواد و فناوری نانو به طور تاریخی از چنین مخازن یکپارچه مدلهای هوش مصنوعی محروم بودهاند.
ماژول AI Core در PyGamLab برای پر کردن این شکاف ایجاد شد.
⚙️ هدف و چشمانداز¶
ماژول ai_core یک چارچوب ساختاریافته و یکپارچه برای
دسترسی، مدیریت و استفاده از مدلهای هوش مصنوعی از پیش آموزش دیده طراحی شده برای
علوم مواد، فیزیک حالت جامد، فناوری نانو و
مهندسی شیمی ارائه میدهد.
به جای صرف هفتهها برای آموزش مجدد مدلهای یادگیری ماشین از
ابتدا — جستجو در GitHub برای پیادهسازیهای جزئی، پردازش مجدد
دادهها، یا ساخت دستی هایپرپارامترها — محققان اکنون میتوانند
بلافاصله مدلهای آماده استفاده ذخیره شده در فرمت .gam_ai را بارگذاری کنند.
این AI Core PyGamLab را نه تنها یک مخزن مدلها، بلکه یک گراف دانش از چشمانداز یادگیری ماشین در علوم مواد میسازد.
🧠 قابلیتهای اصلی¶
ماژول AI Core حول دو مؤلفه کلیدی میچرخد:
.gam_ai شامل موارد زیر است: - دادههای مدل رمزگذاری شده (.joblib
ذخیره شده به صورت Base64)میتوانید به راحتی یک مدل را اینگونه بارگذاری کنید:
from PyGamLab.ai_core import GAM_AI_MODEL
model = GAM_AI_MODEL("cu-nanocomposites-porosity-dt")
model.summary()
این دستور مدل را بارگذاری میکند، متادیتای آن را چاپ میکند (مثلاً اندازه مجموعه داده، منبع آموزش، نویسنده، DOI، و غیره)، و آن را برای پیشبینی آماده میکند. پس از بارگذاری، شیء مدل (model.ml_model) مانند یک تخمینزن scikit-learn عمل میکند — به این معنی که میتوانید مستقیماً فراخوانی کنید:
y_pred = model.ml_model.predict(X_test)
این کلاس یک خط لوله یادگیری ماشین سطح بالا برای مدیریت گردش کارهای end-to-end ارائه میدهد — از بارگذاری مدلها و متادیتا تا انجام پیشبینیها، تولید نمودارها و خلاصهسازی نتایج. مثال استفاده:
from PyGamLab.ai_core import GAM_AI_WORKFLOW
workflow = GAM_AI_WORKFLOW("cu-nanocomposites-porosity-dt")
workflow.get_GAM_AI_MODEL().summary()
این گردش کار به طور یکپارچه با سایر ماژولهای PyGamLab ادغام میشود — مانند Data_Analysis و Structures — که تحقیق بین رشتهای را فعال میکند که در آن میتوانید: پیشبینی خواص مواد (تخلخل، رسانایی، الاستیسیته، و غیره) همبستگی نتایج با مجموعه دادههای تجربی صادرات و نمایش خودکار نتایج
📊 جدول خلاصه ویژگیهای AI Core¶
دسته* |
تابع / کلاس |
توضیحات* |
مثال استفاده |
|---|---|---|---|
Model Access |
|
Load and
manage a
pre-trained
model in
|
|
خودکارسازی گردش کار |
|
خودکارسازی خط لوله ML end-to-end (خلاصه، پیشبینی، نمایش). |
|
خلاصه مدل |
|
نمایش نام مدل، معماری، هایپرپارامترها، منبع و معیارها. |
|
پیشبینی |
|
پیشبینی خواص هدف با استفاده از مدل از پیش آموزش دیده. |
el.predict(X)`` |
نمایش |
` .plot_results()` |
تولید خودکار نمودارها برای دادههای پیشبینی شده در مقابل دادههای واقعی یا اهمیت ویژگی. |
|
دسترسی به متادیتا |
|
بازیابی متادیتای کامل (نویسنده، DOI، عملکرد، و غیره). |
|
ادغام مدل |
|
تبدیل بین مدلهای PyGamLab و Scikit-learn. |
|
ارزیابی |
|
محاسبه معیارهایی مانند MAE، R²، RMSE، و غیره. |
|
اشتراکگذاری مدل |
|
ذخیره یا بارگذاری
مدلهای |
|
سازگاری متقابل* |
ادغام با
|
اتصال پیشبینیهای هوش مصنوعی به مجموعه دادههای مبتنی بر ساختار یا تجربی. |
|
💡 چرا مهم است¶
ماژول AI Core در PyGamLab چیزی بیش از یک ویژگی فنی است — این نمایانگر یک تغییر پارادایم در نحوه رویکرد محققان به هوش مصنوعی در علوم مواد است.
این هفتهها تلاش و قدرت محاسباتی مصرف میکرد.
با AI Core، این فرآیند فوری و شفاف میشود:
🔁 قابلیت تکرار — هر مدل شامل متادیتای نسخهگذاری شده است، که آزمایشها را قابل تکرار و قابل تأیید میسازد.
⚡ سرعت — مدلهای از پیش آموزش دیده پیشبینیهای فوری را فعال میکنند، با رد کردن مراحل جمعآوری داده و آموزش.
🧩 قابلیت همکاری — به طور بومی با سایر ماژولهای PyGamLab کار میکند مانند
data_analysis،structures، وdatabases.🧠 دسترسی — محققان بدون دانش عمیق ML هنوز میتوانند تحلیل پیشرفته و پیشبینی انجام دهند.
🌐 رشد جامعه — اشتراکگذاری و استفاده مجدد از مدلها را در آزمایشگاهها و مؤسسات تشویق میکند و تکرار تلاش را کاهش میدهد.
در نهایت، AI Core شکاف بین نظریه هوش مصنوعی و مهندسی عملی را پر میکند، یادگیری ماشین را به یک ابزار تحقیقاتی روزمره به جای یک چالش تخصصی تبدیل میکند.
🔮 جهتگیریهای آینده¶
AI Core PyGamLab هوش مصنوعی را به یک همراه عملی برای دانشمندان و مهندسان تبدیل میکند.
مزیت |
تأثیر |
|---|---|
مدلهای هوش مصنوعی آماده استفاده |
رد کردن آموزش، شروع به پیشبینی فوری. |
متادیتای استاندارد |
اطمینان از اینکه تحقیق قابل ردیابی و قابل تکرار است. |
سازگاری بین ماژولی |
ترکیب هوش مصنوعی با تحلیل داده و تولید ساختار. |
گردش کارهای مقیاسپذیر |
مدیریت هر دو مجموعه داده کوچک و بزرگ با سهولت. |
همکاری باز |
تشویق اشتراکگذاری و بهبود مدل محور جامعه. |
به طور خلاصه — AI Core جایی است که علوم مواد با هوش ماشین ملاقات میکند.
مثال استفاده¶
#برای شروع گردش کار میتوانید کلاس را import کنید
from PyGamLab.ai_core import Gam_Ai_Workflow
workflow=Gam_Ai_Workflow(model_name='your_model_name_here')
#بنابراین میتوانید فهرست مدلهای موجود را ببینید
#workflow.list_models()
#یا در این وبسایت
#.....
## مثال استفاده
#مثلاً میتوانیم از مدل 'cu-nanocomposites-young-modulus-pipe-mlp' استفاده کنیم
#در واقع ایجاد شیء workflow و استفاده از متدهای آن آسان است
from PyGamLab.ai_core import Gam_Ai_Workflow
workflow=Gam_Ai_Workflow(model_name='cu-nanocomposites-young-modulus-pipe-mlp')
✅ Loaded GAM_AI_MODEL: 'cu-nanocomposites-young-modulus-pipe-mlp'
✅ Loaded model 'cu-nanocomposites-young-modulus-pipe-mlp' (pipe) successfully.
#بنابراین ابتدا میتوانید خلاصه مدل را داشته باشید
workflow.summary()
📘 MODEL SUMMARY
model_name: cu-nanocomposites-young-modulus-pipe-mlp
file_path: /Users/apm/anaconda3/envs/DL/lib/python3.10/site-packages/PyGamLab/ai_core/gam_models/cu-nanocomposites-young-modulus-pipe-mlp.gam_ai
model_type: pipe
description: The presence of vacancy defects in graphene negatively affects the structural behaviors of the composite beams to a certain degree. So, increasing the temperature & defects decrease the mechanical properties, Increasing the percentage of Graphene increase the mechanical properties.
author_name: Shaoyu Zhao, Yingyan Zhang, Yihe Zhang et al.
author_email: None
trainer_name: Ali Pilehvar Meibody
best_accuracy: -0.029005423067057112
doi: https://doi.org/10.1007/s00366-022-01710-w
hyperparam_range: {'model__hidden_layer_sizes': [[50], [100], [100, 50], [100, 100]], 'model__activation': ['relu', 'tanh', 'logistic'], 'model__solver': ['adam', 'lbfgs'], 'model__alpha': [0.0001, 0.001, 0.01], 'model__learning_rate': ['constant', 'adaptive'], 'model__max_iter': [500, 1000]}
best_params: {'model__activation': 'relu', 'model__alpha': 0.01, 'model__hidden_layer_sizes': [100, 100], 'model__learning_rate': 'adaptive', 'model__max_iter': 500, 'model__solver': 'lbfgs'}
ml_model: GridSearchCV(cv=5,
estimator=Pipeline(steps=[('scaler', MinMaxScaler()),
('model', MLPRegressor())]),
n_jobs=-1,
param_grid={'model__activation': ['relu', 'tanh', 'logistic'],
'model__alpha': [0.0001, 0.001, 0.01],
'model__hidden_layer_sizes': [(50,), (100,), (100, 50),
(100, 100)],
'model__learning_rate': ['constant', 'adaptive'],
'model__max_iter': [500, 1000],
'model__solver': ['adam', 'lbfgs']},
return_train_score=True,
scoring='neg_mean_absolute_percentage_error')
#میتوانید ببینید مسیر فایل چیست، نام مدل چیست
#نوع مدل چیست
#توضیحات داده و DOI مقاله
#و بهترین دقت و همچنین مدل
#برای استخراج مدل میتوانید فقط از این متد استفاده کنید
gam_ai_model=workflow.get_GAM_AI_MODEL()
#میتوانید نوع آن را ببینید
print(type(gam_ai_model))
<class 'PyGamLab.ai_core.gam_ai.GAM_AI_MODEL'>
#همچنین کلاس دیگری به نام GAM_AI_MODEL داریم
#برای وارد کردن آن میتوانید از این استفاده کنید
#from PyGamLab.ai_core import GAM_AI_MODEL
#فعلاً فقط summary دارد اما ویژگیهای زیادی دارد
#مثلاً
print('doi:',gam_ai_model.doi)
doi: https://doi.org/10.1007/s00366-022-01710-w
print('description:',gam_ai_model.description)
description: The presence of vacancy defects in graphene negatively affects the structural behaviors of the composite beams to a certain degree. So, increasing the temperature & defects decrease the mechanical properties, Increasing the percentage of Graphene increase the mechanical properties.
#و در نهایت میتوانید فقط مدل sklearn را استخراج کنید با
sklearn_model=gam_ai_model.ml_model
print(type(sklearn_model))
<class 'sklearn.model_selection._search.GridSearchCV'>
#پس برگردیم به کلاس workflow
#در مورد gam_ai_model که از متد workflow.get_GAM_AI_MODEL() است صحبت کردیم
#اکنون میتوانیم از workflow برای ارزیابی عملکرد آن استفاده کنیم
#کافی است از متد evaluate() استفاده کنیم
import numpy as np
x=np.array([[10, 5.0, 0.5],
[20, 7.5, 0.8]])
y=np.array([200.0, 290.0])
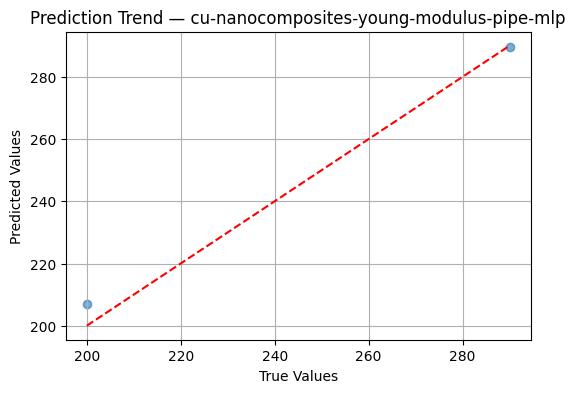
workflow.evaluate_regressor(x,y)
📈 R²: 0.9884
📉 MAE: 3.5745
📉 MSE: 23.4737
#همچنین میتوانید از predict استفاده کنید یا میتوانید از refit استفاده کنید
#اما بهتر است محدوده هایپرپارامتر را ببینید
ضمیمه الف: کار با NumPy¶
🌱 NumPy چیست؟¶
در PyGamLab، NumPy در پشت صحنه برای تقریباً همه چیز استفاده میشود — از مدیریت مختصات ساختار تا انجام محاسبات فیزیکی و تبدیل داده.
🔢 ایجاد آرایهها¶
import numpy as np
# ایجاد آرایههای ساده
a = np.array([1, 2, 3])
b = np.array([[1, 2, 3], [4, 5, 6]])
print(a.shape) # (3,)
print(b.shape) # (2, 3)
همچنین میتوانید آرایههایی با مقادیر از پیش تعریف شده ایجاد کنید:
np.zeros((2, 3)) # آرایه 2x3 پر از صفر
np.ones((3, 3)) # آرایه 3x3 از یکها
np.eye(4) # ماتریس همانی 4x4
np.arange(0, 10, 2) # آرایه از 0 تا 10 با گام 2
np.linspace(0, 1, 5) # 5 مقدار با فاصله مساوی بین 0 و 1
⚙️ عملیات آرایه¶
NumPy عملیات عنصر به عنصر را که هم سریع و هم آسان برای خواندن هستند، امکانپذیر میکند:
x = np.array([1, 2, 3])
y = np.array([10, 20, 30])
print(x + y) # [11 22 33]
print(x * y) # [10 40 90]
print(np.sqrt(y)) # [3.16 4.47 5.47]
🔍 متدهای مفید آرایه¶
a = np.random.rand(5, 5)
print(a.mean()) # میانگین تمام عناصر
print(a.max()) # حداکثر مقدار
print(a.min()) # حداقل مقدار
print(a.sum(axis=0)) # مجموع ستونها
print(a.T) # ترانهاده
🧩 جدول خلاصه: مبانی NumPy¶
مرحله |
وظیفه |
تابع / متد |
توضیحات |
مثال |
|---|---|---|---|---|
1* |
وارد کردن NumPy |
` import numpy as np` |
وارد کردن کتابخانه NumPy |
`` import num py as np`` |
2* |
ایجاد آرایهها |
|
ایجاد آرایهها با اشکال و مقادیر مختلف |
|
3* |
ویژگیهای آرایه |
|
ارائه اطلاعات درباره آرایه |
|
4* |
اندیسگذاری و برش |
|
دسترسی به عناصر یا زیرآرایهها |
`` arr[0:2]`` |
5* |
عملیات ریاضی |
|
انجام عملیات عددی سریع |
|
6* |
پخش |
عملیات ضمنی |
اعمال حساب بین آرایهها با اشکال مختلف |
` arr + 5` |
7* |
اعداد تصادفی |
|
تولید داده تصادفی |
|
8* |
Res hape Arra ys* |
|
Changes the shape of arrays without changing data |
` arr.resha pe(2, 3)` |
9* |
ذخیره و بارگذاری |
|
ذخیره و بارگذاری آرایهها به صورت کارآمد |
|
** 10** |
ادغام با PyGamLab* |
آرایهها به عنوان داده ورودی |
استفاده برای دادههای مواد، مختصات، و شبیهسازیها |
``gam _structure
b.Structur e(np.array ([…]))`` |
یادگیری بیشتر¶
مستندات رسمی NumPy: https://numpy.org/doc روی یادگیری اندیسگذاری، برش و پخش تمرکز کنید — آنها قلب مدیریت کارآمد داده هستند.
ضمیمه ب: کار با Pandas¶
Pandas بهترین دوست شما هنگام کار با دادههای ساختاریافته است — فایلهای CSV، ورقههای Excel، خروجیهای پایگاه داده، یا هر مجموعه داده جدولی.
این روی NumPy ساخته شده و دو ساختار داده اصلی ارائه میدهد: - Series → آرایه برچسبدار یکبعدی (مانند یک ستون) - DataFrame → جدول برچسبدار دوبعدی (مانند یک ورقه Excel)
در PyGamLab، Pandas همه جا استفاده میشود — به ویژه در
ماژولهای data_analysis و databases — برای بارگذاری، پاکسازی و
پردازش دادههای تجربی و شبیهسازی.
🌱 Pandas چیست؟¶
Pandas کار با داده را بسیار آسانتر میکند با امکان: - خواندن/نوشتن سریع فایلها (CSV، Excel، SQL، JSON) - فیلتر کردن آسان داده، گروهبندی و خلاصهسازی - ادغام یکپارچه با NumPy، Matplotlib، و scikit-learn
هرگاه دادههایی در سطرها و ستونها دارید، به Pandas فکر کنید.
📥 خواندن داده¶
import pandas as pd
# خواندن یک فایل CSV
data = pd.read_csv("sample_data.csv")
# خواندن یک فایل Excel
data_excel = pd.read_excel("data.xlsx", sheet_name="Sheet1")
# نمایش پنج سطر اول
print(data.head())
# همچنین میتوانید مستقیماً از یک URL یا اتصال پایگاه داده بارگذاری کنید.
📊 عملیات پایه¶
پس از بارگذاری دادههایتان، میتوانید به راحتی آنها را کاوش و دستکاری کنید:
print(data.columns) # نمایش نام ستونها
print(data.info()) # خلاصه مجموعه داده
print(data.describe()) # آمار پایه برای ستونهای عددی
انتخاب یک ستون یا فیلتر کردن سطرها:
temperatures = data["Temperature"]
filtered = data[data["Pressure"] > 10]
# ایجاد یک ستون محاسبه شده جدید
data["Density_Ratio"] = data["Density"] / data["Temperature"]
تغییر نام ستونها برای وضوح:
data = data.rename(columns={"Temp": "Temperature"})
🔄 ذخیره داده پردازش شده¶
پس از پاکسازی یا تحلیل مجموعه دادهتان، میتوانید آن را دوباره در یک فایل ذخیره کنید:
data.to_csv("processed_data.csv", index=False)
data.to_excel("output.xlsx", sheet_name="Results", index=False)
🧾 جدول خلاصه: Pandas برای مدیریت داده¶
مرحله |
وظیفه |
تابع / متد |
توضیحات |
مثال |
|---|---|---|---|---|
1* |
وارد کردن Pandas |
`` import pandas as pd`` |
وارد کردن کتابخانه Pandas |
|
2* |
ایجاد DataFrame |
|
ایجاد یک ساختار شبیه جدول با سطرها و ستونها |
|
3* |
خواندن داده |
|
بارگذاری داده از فایلهای خارجی |
`` df = pd.re ad_csv(‘da ta.csv’)`` |
4* |
مشاهده داده |
|
نمایش چند سطر اول یا آخر |
|
5* |
بررسی داده* |
|
نمایش ساختار و آمار خلاصه |
|
6* |
انتخاب ستونها / سطرها |
|
دسترسی به زیرمجموعههای داده |
`` df.loc[0, ‘value’]`` |
7* |
فیلتر داده |
اندیسگذاری بولی |
فیلتر بر اساس شرایط |
|
8* |
گروهبندی و تجمیع |
|
گروهبندی داده و محاسبه آمار |
|
9* |
مدیریت مقادیر گمشده |
|
حذف یا جایگزینی دادههای گمشده |
|
** 10** |
صادرات داده |
|
ذخیره داده در فایلها |
|
data_analysis PyGamLab را تشکیل میدهد و برای آمادهسازی ورودیهای مدلهای هوش مصنوعی ضروری است. — ## 📘 یادگیری بیشتر مستندات رسمی:
https://pandas.pydata.org/docs